Vous êtes ici

Océans, sols, intestins… Comment étudier la biodiversité de milieux dont un seul dé à coudre contient des milliards de micro-organismes réfractaires à toute culture en laboratoire ? Réponse : par l’analyse métagénomique. Le concept ? Faire directement parler l’ADN de ces organismes ! À la croisée de la génétique, de l’écologie et de l’informatique, cette discipline émergente est en train de révolutionner de nombreux pans de la recherche.
Une invention aussi importante que le microscope
En métagénomique, la première étape consiste d’abord à extraire l’ADN de l’échantillon prélevé par des traitements physiques, chimiques ou enzymatiques. Cet ADN « en vrac » est alors exploité de diverses manières, en premier lieu à des fins d’inventaire. En effet, certains gènes dits ubiquitaires sont présents chez tous les organismes vivants, par exemple ceux codant pour les ribosomes, mais leur séquence est propre à chaque espèce, constituant ainsi une sorte de code-barres biologique. En amplifiant puis en séquençant ces gènes ubiquitaires, les chercheurs peuvent désormais établir l’inventaire complet des espèces présentes dans l’environnement échantillonné. « C’est une vraie révolution, aussi importante que l’invention du microscope il y a 400 ans… Une sorte de microscope du IIIe millénaire ! », s’enthousiasme Colomban de Vargas, chercheur à la Station biologique de Roscoff1.
plus de deux
millions de clones
contenant chacun
une séquence
d’ADN inconnue
de bactéries du sol.
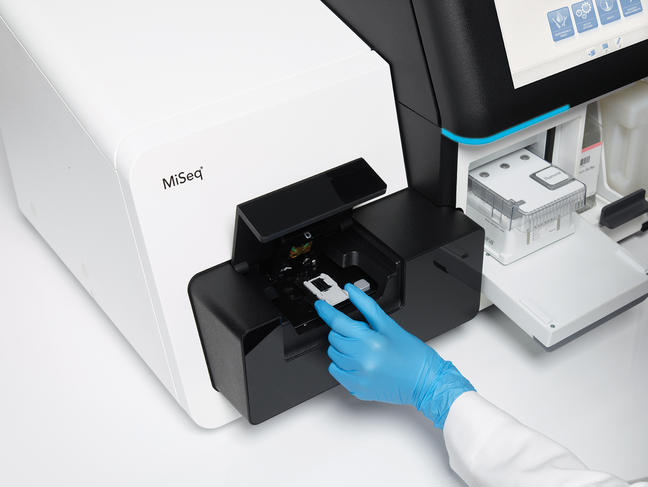
Mais la métagénomique consiste aussi à séquencer la totalité de l’ADN présent. Un séquençage massif et systématique permis grâce au développement récent des techniques du Big Data combiné à l’utilisation de séquenceurs à très haut débit capables de séquencer plusieurs dizaines de milliards de bases nucléiques par jour ! À titre de comparaison, les chromosomes humains n’en contiennent « que » trois milliards… La masse de donnée obtenue après séquençage passe ensuite à la moulinette de logiciels qui peuvent, après avoir identifié certaines séquences parmi d’immenses bases de données génétiques, émettre des hypothèses sur leurs rôles en fonction de similarités avec des séquences connues, déterminer les interactions entre les divers organismes identifiés, etc.
On peut même insérer certaines séquences génétiques inconnues dans le génome de bactéries afin de les faire « s’exprimer », et ainsi découvrir leurs fonctions en tant que gènes. « Dans notre laboratoire, nous avons ainsi créé plus de deux millions de clones contenant chacun une séquence d’ADN inconnue de bactéries du sol », explique Pascal Simonet, chercheur au laboratoire Ampère de l’École centrale de Lyon2.
Zoom sur la biodiversité aquatique
Combinés, tous ces outils ont fait faire des bonds de géant à la biologie. Exemple : la récente analyse métagénomique de 579 échantillons d’eau de mer récoltés aux quatre coins du globe par la mission Tara Oceans. Objectif : étudier la biodiversité du plancton, cette myriade de micro-organismes à la base de la chaîne alimentaire océanique et qui produit près de 50 % de l’oxygène atmosphérique. Ainsi, au total, l’équipe Tara a séquencé une quantité d’ADN planctonique équivalant à près de 2 000 génomes humains, identifiant près de 40 millions de gènes différents, dont 80 % jusqu’ici inconnus ! « Rien que pour les organismes dotés d’un noyau (eucaryotes), nous avons séquencé près d’un milliard de code-barres génétiques, indique Colomban de Vargas. Nous avons ainsi mis à jour une diversité d’organismes unicellulaires (protistes) bien plus grande que prévu. » Les chercheurs de Tara ont également collecté le matériel génétique de plus de 35 000 espèces de bactéries planctoniques différentes. Grâce à de nouveaux modèles informatiques, ils ont même établi la nature des interactions, souvent parasitiques, entre virus, bactéries et eucaryotes.
jour une diversité
d’organismes
unicellulaires
(protistes)
bien plus grande
que prévu.
Appliquée aux grands fonds marins, la métagénomique fait aussi des merveilles. Ainsi, en mai dernier, des chercheurs suédois ont fait une découverte cruciale publiée dans la célèbre revue Nature. Par analyse métagénomique des sédiments du château de Loki – une source hydrothermale située à plus de 2 300 mètres de fond entre le Groenland et la Norvège – équipe a identifié un tout nouveau groupe d’archées, des micro-organismes unicellulaires différents des bactéries et des eucaryotes. Leur particularité ? Ils possédaient de nombreux gènes de type eucaryotes.
Depuis longtemps, les spécialistes suspectaient les archées d’avoir été les ancêtres des eucaryotes, et donc de toutes les plantes et tous les animaux. Nommé Lokiarchaeota, ce nouveau groupe semble bien être le chaînon qui manquait pour expliquer l’apparition des eucaryotes il y a plusieurs milliards d’années ! Les écosystèmes océaniques ne sont d’ailleurs pas seuls à receler une étonnante diversité microbienne. « Depuis quelques années, l’étude métagénomique des lacs révèle aussi une extrême diversité génétique de virus avec de très nombreux gènes jamais vus auparavant », complète Francois Enault, bio-informaticien spécialiste des virus aquatiques au laboratoire clermontois Micro-organismes : génome et environnement3.

Du monde sous nos pieds… et dans nos intestins
Un autre gisement de biodiversité microbienne se cache aussi sous nos pieds. Un seul gramme de terre peut en effet contenir jusqu’à un milliard de bactéries ! Et là encore, seule la métagénomique est capable d’étudier ce colossal réservoir biologique souterrain, qui regorge notamment de bactéries productrices d’antibiotiques naturels. « Alors que certaines bactéries pathogènes pour l’homme résistent aujourd’hui à tous les antibiotiques disponibles, ce gisement de nouveaux antibiotiques mis à jour par métagénomique est une excellente nouvelle », se félicite Pascal Simonet. Mais les antibiotiques sont loin de constituer la seule application potentielle de l’étude métagénomique des bactéries du sol. L’idée est aussi de trouver de nouvelles enzymes utiles pour l’industrie ou l’agriculture, mais aussi pour dépolluer ; c’est ainsi que l’on a découvert une bactérie dotée d’un gène capable de dégrader le lindane, un pesticide extrêmement persistant.
de nouveaux
antibiotiques
découvert par
métagénomique
est une excellente
nouvelle.
Enfin, l’analyse métagénomique a aussi été appliquée au corps humain, notamment à notre intestin. En effet, ce dernier recèle près de 100 000 milliards de micro-organismes, soit dix fois plus que nos propres cellules ! Une biodiversité jusque-là méconnue, car la plupart des espèces hébergées par notre système digestif ne sont pas cultivables in vitro. « Les analyses métagénomiques ont révélé une grande variabilité de familles bactériennes intestinales articulée autour de trois grands types de compositions dits entérotypes, indique Pascal Simonet. Quant à la part de flore intestinale propre à chaque individu, elle semble moins importante que prévu. »
En décryptant les fonctions de ce « second génome », les chercheurs ont aussi découvert qu’une flore intestinale saine est indispensable au bon fonctionnement de la digestion bien sûr, mais aussi pour le métabolisme, l’immunité… ou bien encore pour le système nerveux. Obésité, diabète, maladies cardiovasculaires, allergies, maladies inflammatoires : les déséquilibres de la flore intestinale sont aujourd’hui suspectés d’être à l’origine d’une kyrielle de pathologies.
Médecine, protection de l’environnement, paléontologie, agroalimentaire… Si la métagénomique reste avant tout utilisée pour mieux connaître la biodiversité microbienne, ses potentielles applications intéressent de plus en plus de secteurs. Cette discipline en plein boom n’a pas fini de nous surprendre…
Mots-clés
Partager cet article
Auteur
Journaliste scientifique, Jean-Philippe Braly est spécialisés dans les thématiques santé/sciences du vivant, environnement/écologie/nature et nouvelles technologies. Il collabore notamment avec CNRS Le journal, le magazine La Recherche, les agences de presse Canopy et Look at Sciences. Producteur de documentaires pour l’émission de France Culture « Sur les Docks », il anime...







































































































Commentaires
Connectez-vous, rejoignez la communauté
du journal CNRS