Vous êtes ici
Contrer l’antibiorésistance
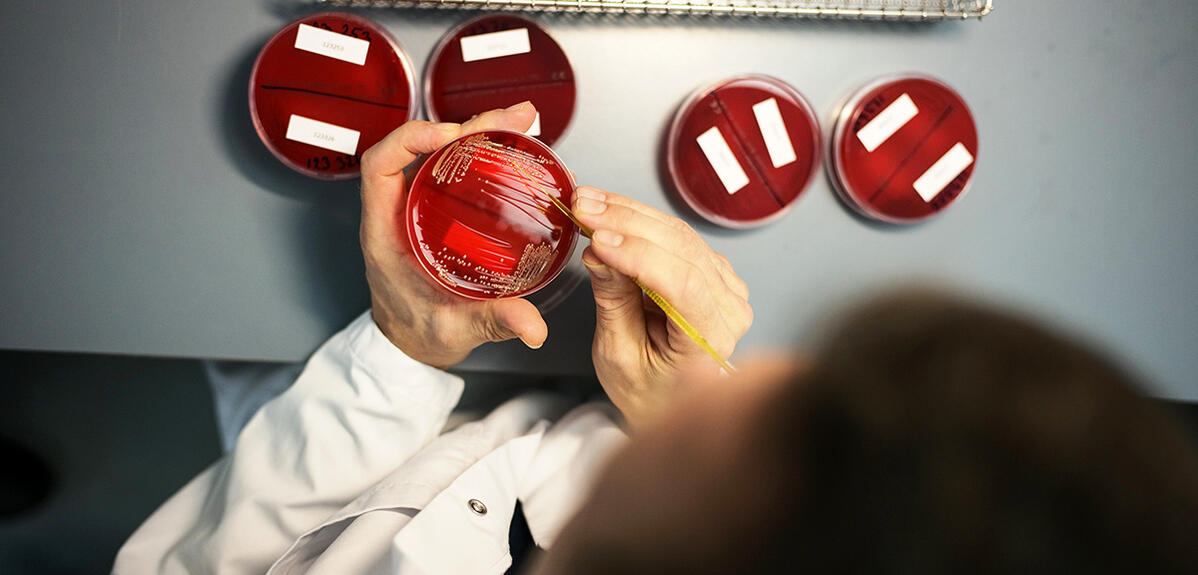
Le constat est unanime : si rien n’est fait, la résistance des pathogènes aux agents antimicrobiens pourrait devenir la première cause de mortalité dans le monde à l’horizon 2050. Comme l’affirmait l’Organisation mondiale de la santé (OMS) dès 2015, le phénomène « représente un immense danger pour la santé mondiale » pouvant conduire à une « ère post-antibiotique dans laquelle les infections courantes pourront recommencer à tuer ». Comme un retour à l’époque d’avant la découverte de la pénicilline… Conscients du danger, les scientifiques du monde entier ont pris le problème à bras-le-corps. Biologistes et chimistes sont en première ligne et, avec eux, les experts du CNRS et de ses partenaires.
L’acquisition de résistances aux antimicrobiens, des médicaments utilisés pour prévenir et traiter les infections, est un phénomène évolutif naturel. Au cours du temps, les pathogènes évoluent, la sélection naturelle favorisant les micro-organismes les mieux adaptés à leur environnement, ici en les rendant insensibles aux médicaments auxquels ils sont exposés. Mais rien que ces dernières décennies, une utilisation parfois abusive et excessive des antibiotiques a fait prendre au phénomène une tournure nous plaçant dans une situation critique.

Concrètement, on observe désormais un taux élevé de résistance pour des infections bactériennes courantes. Avec des cas de propagation mondiale rapide de pathogènes dits « multi-résistants » qui, pour certains d’entre eux, ne peuvent tout simplement plus être traités via les traitements existants. Alors que depuis près d’un siècle l’essor des antibiotiques nous avait laissé croire que le risque infectieux était sous contrôle, nous sommes tenus de réapprendre dans l’urgence que la lutte contre les infections ne peut être que sans relâche. Elle implique une compréhension fine des processus par lesquels un microbe acquiert des résistances, comment celles-ci se diffusent, et demande une innovation permanente en matière de conception de diagnostics, de molécules antimicrobiennes et de définition de stratégies thérapeutiques.
Comprendre les différentes causes de résistance
Comme l’explique Olivier Neyrolles, à l’Institut de pharmacologie et de biologie structurale1 (IPBS) à Toulouse, « la résistance d’un pathogène à un antimicrobien peut avoir différentes origines. Elle peut être liée à une mutation de la cible qui n’est alors plus reconnue par le médicament, ou bien à une modification d’une enzyme nécessaire à l’activation de ce dernier. Parfois c’est la membrane du pathogène qui est rendue imperméable aux médicaments ou alors c’est la capacité du pathogène à excréter le médicament qui est accrue. »
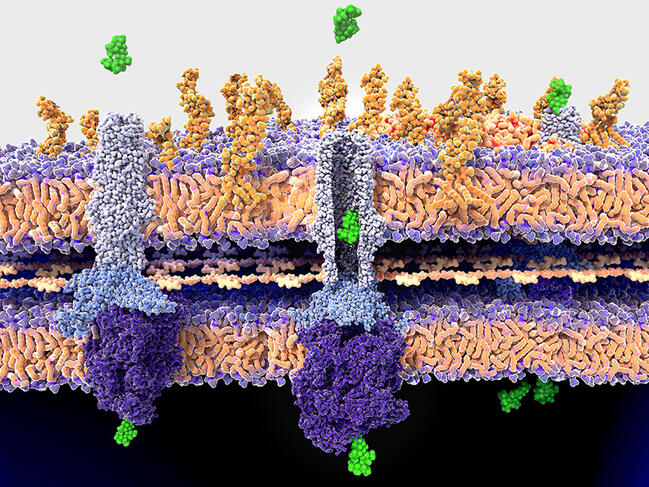

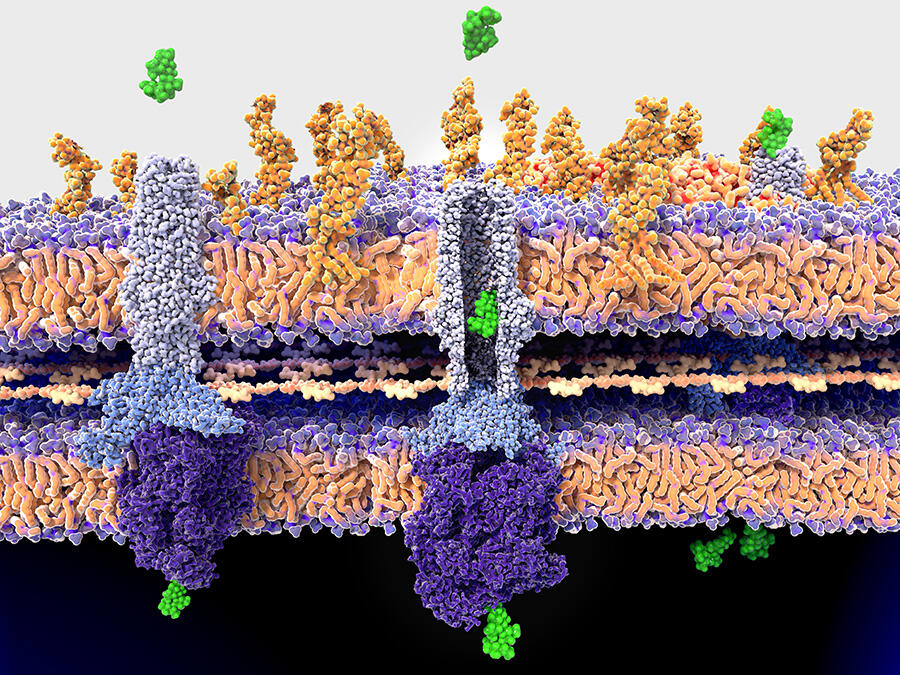
Ainsi, la première mission des spécialistes consiste-t-elle à comprendre le plus finement possible les voies multiples empruntées par la résistance, et la manière dont surviennent et se diffusent les mutations génétiques qui en sont à l’origine. Par exemple, récemment, Philip Supply, au Centre d’infection et d’immunité de Lille2, a déterminé les facteurs qui ont permis l’émergence et la propagation rapide de la branche épidémique dite W148 d’une lignée de souches de la tuberculose. Particulièrement virulente, les souches de W148 résistent également jusqu’à onze médicaments. Résultat : non seulement ce variant du pathogène présente de multiples mutations de résistance, mais également des mutations permettant de compenser le coût métabolique de leur acquisition, et enfin une mutation d’hypervirulence. « Nos données montrent que cette dernière préexiste aux mutations de résistance. Et c’est la combinaison de l’ensemble qui fait la dangerosité de ce variant », précise le scientifique.
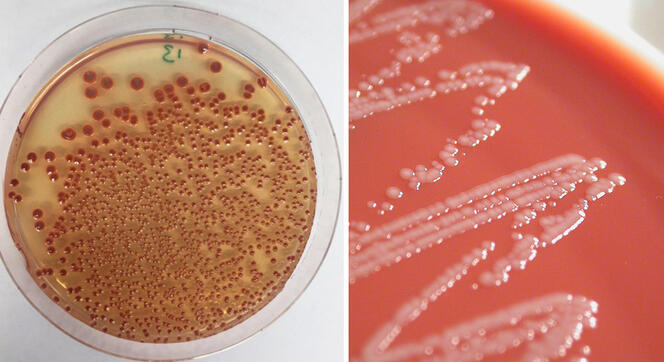
Dans certains cas, on observe également des transferts de gènes entre micro-organismes, y compris d’espèces différentes. C’est notamment le cas de Acinetobacter baumannii, une bactérie responsable d’infections opportunistes et nosocomiales difficilement traitables, et classée par l’OMS parmi les souches prioritaires à étudier pour la recherche de nouveaux antibiotiques. L’année dernière, Xavier Charpentier, au Centre international de recherche en infectiologie3, à Lyon, a en effet montré que des souches de cette bactérie a priori sensibles à un antibiotique sont capables en quelques heures d’acquérir des résistances. Comment ? Par un processus de captage et d’incorporation d’une partie du génome de ses voisines. « On ne comprend pas encore très bien les conditions qui président à ce transfert, mais nous avons observé que jusqu’à 80 nouveaux gènes peuvent être acquis par une bactérie lors d’un seul événement de transfert, ce qui en fait un processus d’une redoutable efficacité », précise le biologiste.
Adapter les traitements
« L’objectif de la communauté scientifique est désormais de cartographier de manière globale la résistance, à travers des analyses génomiques, d’expression, protéomiques et métabolomiquesFermerLa métabolomique est l'étude de l'ensemble des métabolites (petites molécules) présents dans un organite, une cellule, un tissu, un organe ou un organisme à un temps donné et dans des conditions données », commente Paola Arimondo, à la tête de l’équipe Chimie biologique épigénétique, au laboratoire Chimie biologique pour le vivant4 (Chem4Life). « Cela permettra d’anticiper les résistances qui pourraient survenir, afin d’adapter les traitements en fonction du type de résistances auxquelles on a à faire face, que ce soit à l’échelle du patient ou au niveau national, mais aussi pour réglementer l’usage des antibiotiques en santé humaine, animale et agricole », ajoute Xavier Charpentier.
Dans ce but, Philip Supply, en collaboration avec l’entreprise GenoScreen, a mis au point des nouveaux diagnostics moléculaires pour la lèpre et la tuberculose. Fondé sur la technique de séquençage ciblé en profondeur, ils permettent, à partir d’un prélèvement de patient et sans mise en culture, de déterminer les résistances de la souche en présence et par conséquent d’adapter au mieux le traitement. « Dans le cas de la tuberculose, notre test, le plus complet à ce jour, permet d’établir simultanément la sensibilité ou la résistance d’une souche à 13 classes d’antibiotiques », détaille le chercheur. Après une déclaration d’invention déposée en 2018, un contrat de licence a été signé en 2021 avec GenoScreen pour l’exploitation de la découverte. Et l’entreprise lilloise vient d’annoncer la signature d’un partenariat avec la firme Illumina, leader mondial du séquençage.

Au-delà de notre capacité à diagnostiquer et suivre l’évolution de mutations, la question du traitement des infections demeure cruciale. Et face à la diminution de l’efficacité des antibiotiques, tous nos schémas sont à revoir. En modifiant les cocktails d’antibiotiques en usage, mais aussi en concevant de nouvelles drogues. C’est par exemple ce à quoi s’emploie Jean-François Hernandez, à l’Institut des biomolécules Max Mousseron5, à Montpellier. Précisément, ce chimiste travaille à proposer des molécules capables de contrer certains modes de résistances des bactéries dites à « Gram négatif » aux antibiotiques de la famille des carbapénèmes. Ceux-ci sont utilisés exclusivement à l’hôpital comme dernier recours, et la problématique de la résistance à leur effet a été classée ultra-prioritaire par l’OMS.
Contrer les mécanismes d'adaptation des pathogènes
Parmi les multiples adaptations de ces pathogènes, l’une d’elles tient à l’expression d’une famille d’enzymes appelées métallo-bêta-lactamases, capables d’inactiver les carbapénèmes. Cette famille a émergé il y a une trentaine d’années et s’est largement répandue depuis, sans qu’il existe aujourd’hui d’inhibiteur de ces enzymes sur le marché. À la suite d’une observation inattendue faite en 2006 au sein de l’équipe d’Otto Dideberg, à l’Institut de biologie structurale6 (IBS) de Grenoble, suivie d’un patient travail d’optimisation, Jean-François Hernandez et ses collaborateurs sont parvenus à mettre au point une série de molécules capables d’inhiber l’action de ces métallo-bêta-lactamases. Un brevet a été déposé en 2018, et un programme de pré-maturation a été financé par la Société d’accélération de transfert technologique AxLR. « In vitro et in vivo, nous avons montré que nos molécules sont capables de restaurer l’effet d’un carbapénème sur certaines souches cliniques productrices de ces enzymes et particulièrement problématiques, explique le chercheur. C’est très prometteur et nous cherchons désormais des partenaires pour aller plus loin. »


Parmi les différentes stratégies actuellement à l’étude, l’une consiste à chercher à cibler des processus dits « non essentiels » d’un pathogène. Comme l’explique Olivier Neyrolles, « on entend par là des processus qui inhibés in vitro n’empêchent pas le pathogène de proliférer, mais qui in vivo sont cruciaux à un moment précis de son cycle de vie. » L’intérêt : parce que moins vitaux pour l’agent pathogène, ces processus sont moins soumis à la pression de sélection. Leur ciblage ne devrait donc pas engendrer l’apparition trop rapide de résistances.
À l’IPBS de Toulouse, plusieurs équipes travaillent dans ce sens afin de proposer de nouveaux traitements contre la tuberculose. L’OMS estime que près d’un demi-million de nouveaux cas de cette maladie résistants à l’antibiotique principal de première intention se produisent chaque année. Ainsi, l’une de ces équipes a identifié une enzyme qui participe à la synthèse des lipides de l’enveloppe du bacille. « Lorsqu’il en est privé in vitro, le bacille n’en est pas affecté. En revanche, à une étape du cycle infectieux, son ciblage est délétère », précise Olivier Neyrolles.



À la suite de cette observation, les scientifiques ont identifié et validé des composés capables de déstabiliser ce complexe enzymatique. Un brevet a été déposé en 2022 et des tests in vivo sont programmés pour améliorer les aspects de pharmacocinétique. En parallèle, une autre équipe du laboratoire a récemment démarré un programme pour trouver des molécules susceptibles de s’attaquer à l’enzyme PMT, elle aussi impliqués dans le cycle infectieux de la tuberculose. Enfin, dans une perspective très amont, une troisième équipe cherche à déstabiliser un système toxine/antitoxine propre au bacille dans la perspective qu’il s’autodétruise.
S'intéresser aux processus épigénétiques
De leur côté, les scientifiques du laboratoire Chem4Life s’intéressent à la possibilité de tirer parti de processus épigénétiques dans la lutte contre les infections résistantes. Comme l’explique Paola Arimondo, « l’épigénétique concerne des modifications chimiques réversibles de l’ADN et les protéines autour desquelles l’ADN s’enroule, qui font que dans des cellules de natures différentes, ou des environnements différents, un même génome ne s’exprime pas de la même manière. » Ainsi, à différents moments de son cycle infectieux, un même pathogène va connaître des modifications épigénétiques de son ADN afin d’adapter son fonctionnement aux différents environnements auxquels il est confronté (réservoir animal, différentes cellules de l’hôte…). D’où l’idée de s’attaquer aux protéines impliquées dans ces modifications afin de perturber le cycle de vie d’un agent infectieux.
Concrètement, en 2018, l’équipe de la chercheuse et celle d’Artur Scherf, de l’unité Biologie des interactions hôte-parasite7, à l’Institut Pasteur, ont découvert plusieurs séries de molécules capables d’inhiber les protéines associées aux modifications épigénétiques du paludisme. « Des tests ont montré que ces molécules sont actives sur une cinquantaine de souches résistantes, précise Paola Arimondo. Nous avons déposé un brevet en 2021 et nous sommes désormais à la recherche de partenaires industriels pour aller plus loin. »
À plus long terme, ces chercheurs, dans un projet impliquant huit équipes de recherche, envisagent également de cibler des protéines de sept bactéries, dont le rôle est de modifier l’épigénome de l’hôte pour proliférer et se développer, par exemple en échappant à la réponse immunitaire de l’organisme contaminé. En complément d’antimicrobiens plus classiques, une telle approche pourrait elle aussi offrir des solutions de lutte contre les résistances.
Utiliser des microbes pour vaincre d'autres microbes
Une chose est sûre, alors qu’en 2020 l’OMS déplorait que le développement clinique de nouveaux antimicrobiens était au point mort, toutes les pistes se doivent d’être explorées. L’une d’elles, empruntée par Véronique Éparvier, à l’Institut de chimie des substances naturelles8, consiste à rechercher des solutions parmi celles directement proposées par le vivant. Spécialiste des symbioses entre micro-organismes et animaux ou végétaux, cette chimiste est à l’origine de la souchothèque du CNRS. Cette collection regroupe environ 2 000 souches microbiennes qui constituent une ressource inépuisable de dizaines de milliers de molécules dont certaines ont un effet antibiotique potentiellement exploitable pour la médecine humaine.

Dans le cadre de collaborations avec le Centre national de référence de la résistance aux antibiotiques, la scientifique a isolé des molécules antimicrobiennes produites par des champignons symbiotiques des termites. Comme elle l’explique, « nos premiers tests montrent que ces molécules sont actives contre des champignons du genre trichophytons, responsables de dermatophytose, mais aussi contre le staphylocoque doré résistant à la méticilline. » Par ailleurs, quoique non encore testés sur des pathogènes humains, Véronique Éparvier a également isolé des cyclopeptides synthétisés par des endophytesFermerChampignons se développant à l'intérieur de végétaux. d’une espèce de palmier, efficaces contre certains pathogènes des plantes.
Prudente, Paola Arimondo insiste : « il ne faudrait pas laisser croire que nous sommes arrivés. On parle ici de recherches de longue haleine et qui nécessiteront beaucoup de travail et d’investissement. » « Je pense que la prise de conscience du problème a eu lieu au niveau institutionnel et académique », complète, optimiste, Philip Supply. C’est heureux, car contre la résistance aux antibiotiques, c’est une certitude, le chronomètre est enclenché. ♦
À lire sur notre site
Ces bactéries qui vous veulent du bien
- 1. Unité CNRS/Université Toulouse Paul Sabatier.
- 2. Unité CNRS/CHU de Lille/Inserm/Institut Pasteur de Lille/Université de Lille.
- 3. Unité CNRS/ENS Lyon/Inserm/Université Claude Bernard.
- 4. Unité CNRS/Institut Pasteur.
- 5. Unité CNRS/École nationale supérieure de chimie de Montpellier/Université de Montpellier.
- 6. Unité CNRS/CEA/Université Grenoble-Alpes.
- 7. Équipe mixte de recherche Bases génétiques et moléculaires des interactions hôte-parasite (unité CNRS/Inserm/Institut Pasteur).
- 8. Unité CNRS/Université Paris-Saclay.
Mots-clés
Partager cet article
Auteur
Né en 1974, Mathieu Grousson est journaliste scientifique. Diplômé de l’École supérieure de journalisme de Lille, il est également docteur en physique.

















Commentaires
Article très intéressant,
laurent pagani le 5 Mai 2023 à 09h47Connectez-vous, rejoignez la communauté
du journal CNRS