A la une
Dans l’ensemble du monde vivant, des ions métalliques confèrent aux protéines des fonctions spéciales. Des chercheurs en biologie structurale sont parvenus à élucider le rôle d’interrupteur génétique de centres métalliques fer-soufre dans des protéines bactériennes.
Pour un micro-organisme tel qu’une bactérie, pouvoir réagir pour s’adapter rapidement aux perturbations de son environnement, est un processus absolument nécessaire à sa survie. Pour ce faire, elle dispose de protéines régulatrices, appelées « facteurs de transcription », qui peuvent intervenir au niveau de la lecture de l’ADN, afin de bloquer ou favoriser la synthèse d’autres protéines dont la fonction permet l’adaptation aux changements.
Pour assurer leur rôle, certaines protéines font intervenir un centre métallique, on parle alors de métalloprotéines. Le projet de recherche MANGO-ICING[1], porté par des chercheurs de l’Institut de biologie structurale (IBS), vise à élucider le rôle de plusieurs facteurs de transcription bactériens portant un assemblage d’atomes de fer et de soufre, appelé centre fer-soufre (Fe-S). Chez de multiples espèces bactériennes, ces centres sont impliqués dans la réponse à des perturbations environnementales diverses.
Des facteurs de transcription réceptifs aux signaux environnementaux
Comment des agrégats métalliques peuvent-ils capter des signaux environnementaux et jouer le rôle crucial d’interrupteur de la transcription génétique, provoquant des changements conformationnels dans la protéine régulatrice ? Pour répondre à cette question, les chercheurs de l’IBS se sont focalisés sur quatre facteurs de transcription (FT) à centre Fe-S, appartenant à une famille de protéines spécifique aux bactéries. Très proches structuralement, ces 4 FTs répondent pourtant à des signaux bien distincts, se fixant sur des séquences d’ADN particulières, afin de contrôler :
• La concentration cellulaire en fer (par le capteur de Fe RirA) ;
• La réponse au stress induit par le monoxyde d’azote (par le capteur de NO NsrR) ;
• L’assemblage des centres Fe-S (par le capteur de Fe et S IscR) ;
• L’équilibre d’oxydo-réduction de la cellule (par le capteur d’un excès d’électrons RsrR).
Quelles transformations de la structure du centre Fe-S provoquée par ces signaux peuvent expliquer de telles différences fonctionnelles pour la protéine, puis pour la bactérie ?
Piloté par Anne Volbeda, Juan Carlos Fontecilla-Camps et Eve de Rosny, le projet MANGO-ICING a pu compter sur une équipe multidisciplinaire d’une dizaine de personnes, en partenariat avec le groupe de recherche britannique de Nick Le Brun, de l’Université d’East Anglia, à Norwich. De nombreuses compétences et spécialités scientifiques ont été mobilisées : cristallographie, calculs théoriques, biochimie, ou encore spectroscopie.
Observer les métalloprotéines ou les défis de la cristallographie aux rayons X
Chaque protéine ayant une structure déterminante pour sa fonction biologique, l’équipe MANGO-ICING devait parvenir à caractériser l’agencement des atomes en trois dimensions de chacun des 4 FTs. Les étapes de production, de purification et enfin de cristallisation de ces métalloprotéines ont nécessité de délicates expérimentations dans un environnement sans oxygène, parce qu’elles y sont particulièrement sensibles. Ces conditions anaérobies ont été assurées par des « boîtes à gants » (figure 1).[2]

Figure 1: Manipulation des métalloprotéines sensibles à l’oxygène dans les « boîtes à gants » (BAG). L’équipe Métalloprotéines de l’IBS dispose d’une salle équipée de 6 BAGs, dont certaines sont équipées de robots qui simplifient considérablement le travail des cristallographes. Ce sont notamment : a, une station de pipetage, qui réalise automatiquement la préparation de nanogouttes de cristallisation dans des plaques, en utilisant des milliers de conditions avec des propriétés physico-chimiques différentes ; b, un bras robotique qui déplace les plaques de cristallisation pour les poser sous un appareil photo, afin de suivre l’apparition des cristaux au cours du temps. Souvent, dans une autre BAG, il est nécessaire de modifier, manuellement, les conditions de cristallisation pour obtenir des meilleurs cristaux. Dans la photo, on voit une des BAGs utilisées pour purifier une protéine sensible à l’O2. ©Eve de Rosny – IBS (CEA-CNRS-UGA)
Avant d’exposer la métalloprotéine à un faisceau de rayons X très puissant, un défi majeur s’impose : parvenir à la cristalliser. Eve de Rosny insiste sur le caractère empirique dans la recherche des conditions physico-chimiques de cristallisation pour chaque FT.
« Il n’y a pas de recette miracle, on teste différents sels, des agents précipitants, on modifie le pH… et de temps en temps une condition va permettre aux protéines de cristalliser, mais on ne peut pas prédire laquelle »
Des milliers de conditions ont été testées pour tenter de cristalliser les 4 FTs choisis. Iscr et RirA[3] ne se sont pas laissés cristalliser malgré plus de 3 600 conditions testées, contrairement à RsrR (figure 2A) et NsrR qui ont donné lieu à des cristaux exploitables.

Figure 2. Collection de données de diffraction d’un cristal. A) Cristaux de la forme réduite de la RsrR (taille environ 0.1 mm), obtenus en conditions d’anaérobie dans une BAG. ©Anne Volbeda – IBS (CEA-CNRS-UGA). B) Utilisation d’un faisceau très intense de rayons X (longueur d’onde fixe, réglable autour de 1 Å) généré par le synchrotron à Grenoble. ©ESRF/Jocelyn Chavit. C) Exemple d’un cliché de diffraction aux rayons X pour le cristal d’une protéine. Des milliers de clichés sont obtenus en tournant le cristal autour d’un axe perpendiculaire au faisceau de rayons X. Sur ces images, on détermine la distribution et l’intensité des tâches de diffraction qui dépendent de l’agencement des atomes dans le cristal. ©Anne Volbeda – IBS (CEA-CNRS-UGA)
Les précieux cristaux sont ensuite péchés avec des petites boucles et congelés dans la boîte à gants à -173 °C pour empêcher leur réaction avec l’oxygène ; ils peuvent alors être sortis et transportés dans l’azote liquide (-190 °C) jusqu’au synchrotron européen de Grenoble (figure 2B) à côté de l’IBS, pour être exposés aux rayons X, aussi à une très basse température d’environ -173 °C.
Il faut ensuite toute l’expertise et la patience du cristallographe, pour interpréter l’ensemble des données de diffraction obtenues grâce aux rayons X du synchrotron (figure 2C) et en déduire la structure en trois dimensions de la protéine (figure 3).
Anne Volbeda partage avec nous son enthousiasme de parvenir alors à « […] voir des choses que personne n’avait encore jamais vues. Les structures des protéines sont esthétiquement très belles. »
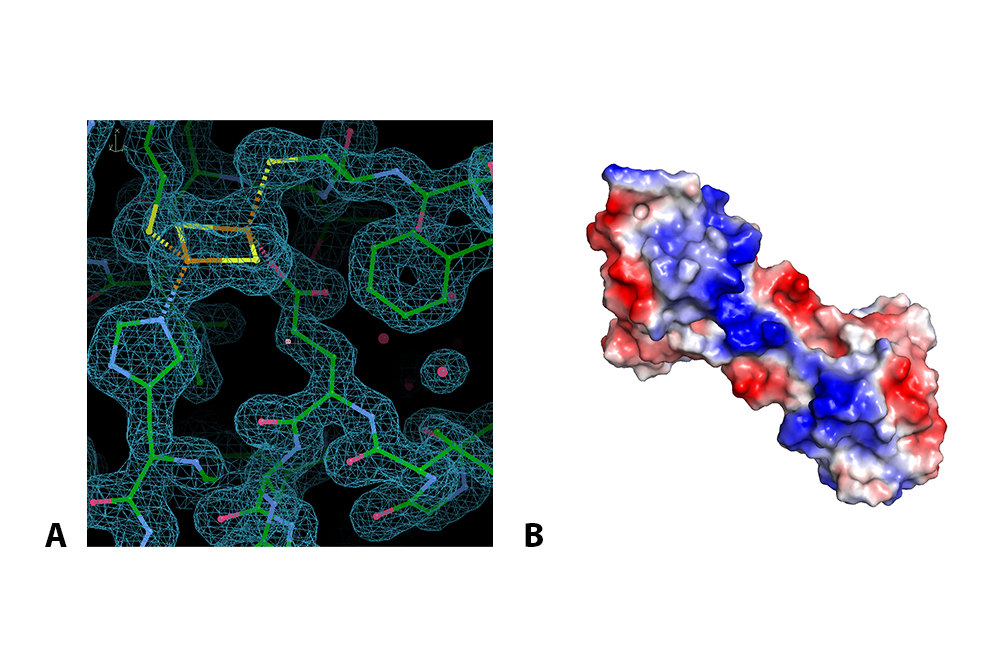
Figure 3. Résolution de la structure. À partir des images de diffraction, on construit un jeu de données expérimentales qui rassemble les amplitudes de toutes les ondes de rayons X diffractées par le cristal. A) Ensuite il faut de nombreux calculs pour obtenir les phases des ondes, et arriver à générer une carte de densité électronique. À partir de cette carte, le chercheur place les atomes et les liaisons qui les relient, en utilisant un écran graphique et des logiciels très performants. Dans la figure A, on voit le centre [2Fe-2S] de RsrR avec les liaisons chimiques fer-soufre en marron et jaune. Enfin, il parvient à la modélisation complète de la structure tridimensionnelle. B) Il y a plusieurs façons de représenter la structure d’une protéine – ici on voit une représentation de la surface de RsrR : les couleurs rouge et bleu montrent les surfaces avec des charges, respectivement, négatives et positives. Ensuite, il reste à résoudre la question la plus intéressante : comment fonctionne la protéine ? On parle de « relations structure-fonction ». ©Anne Volbeda – IBS (CEA-CNRS-UGA)
Élucider les liens entre la structure et la fonction du centre fer-soufre
La structure en 3D de la métalloprotéine permet aux chercheurs d’observer le centre Fe-S dans son environnement protéique. Il s’agit de comprendre comment un petit changement de structure de ce centre conduit à des changements majeurs dans le fonctionnement de la cellule.
Anne Volbeda nous explique le lien intime entre la structure et la fonction du centre fer-soufre : « C’est le centre fer-soufre qui capte le signal de l’environnement ; ensuite c’est l’interaction entre le centre et la protéine qui devient important. Le centre change sa conformation : il perd du fer, ou bien il capte ou perd un électron. Ces changements conformationnels dans l’environnement protéique, ont des conséquences pour l’affinité de la protéine pour l’ADN. »
Eve de Rosny précise qu’au sein de ces protéines, le centre Fe-S ne se trouve pas à l’endroit qui interagit avec l’ADN.
« Il y a une transmission de signal, depuis la zone de la protéine dans laquelle se trouve le centre, vers une autre zone de la protéine qui va changer de forme et conduire la protéine à ne plus reconnaître l’ADN. »
C’est donc une véritable cascade de changements structuraux dans la protéine, induite par la réaction chimique au niveau du centre Fe-S, qu’il s’agit pour les chercheurs de parvenir à retracer.
Le projet MANGO-ICING aura notamment permis de décrire structuralement le rôle d’interrupteur génétique d’un centre Fe-S, pour deux FTs.
RsrR ou les effets en cascade provoqués par un unique électron
La résolution de la structure de la métalloprotéine bactérienne RsrR[4] (Figure 3) accompagnée par plusieurs autres analyses[5] a permis de montrer comment un simple électron pouvait moduler la capacité de fixation à l’ADN de ce FT. La capture de l’électron au centre [2Fe-2S]2+ cause l’addition d’une charge positive dans son environnent proche, provoquant ainsi un réarrangement structural qui modifie la surface de la protéine et réduit ainsi ses capacités à se fixer à l’ADN.
NsrR ou comment contourner les défenses immunitaires de son hôte
Le projet MANGO-ICING a également permis de mieux comprendre comment le centre [4Fe-4S] au sein du FT NsrR[6], permet à une bactérie pathogénique de résister à l’une des armes immunitaires de son hôte : le monoxyde d’azote (NO). La production de ce gaz par les macrophages est un moyen de défense courant pour les organismes infectés par des bactéries. La NsrR permet à la bactérie de s’adapter et de résister à cette réponse en neutralisant le NO.
Eve de Rosny et Anne Volbeda insistent sur le caractère fondamental de leur recherche. Les applications de leurs travaux ne peuvent être imaginées que sur le plus long terme. Il s’agit pour les chercheurs d’apporter leur pierre, ou plutôt leur cristal, à la déjà vaste « encyclopédie des connaissances ».
_ _ _
Ces recherches ont été financées en tout ou partie, par l’Agence nationale de la recherche (ANR) au titre du projet ANR-MANGO-ICING-AAPG2018. Cette communication est réalisée et financée dans le cadre de l’appel à projet Sciences Avec et Pour la Société - Culture Scientifique Technique et Industrielle pour les projets JCJC et PRC des appels à projets génériques 2018-2019 (SAPS-CSTI-JCJC et PRC AAPG 18/19).
_ _ _
[1] MANGO-ICING : Mechanisms of gene transcription regulation through iron-sulfur cluster signaling
[2] Le laboratoire de pointe de l’IBS, spécialisé dans les expérimentations en conditions sans oxygène, a été imaginé par Juan C. Fontecilla-Camps, bien avant l’émergence du projet MANGO-ICING. Il est l’une des raisons d’être de l’Institut de biologie structurale de Grenoble, créé conjointement par le Commissariat à l’énergie atomique et aux énergies alternatives (CEA) et le CNRS en janvier 1992.
[3] En partenariat avec l’équipe britannique de Nick E. Le Brun, Anne Volbeda et Juan C. Fontecilla-Camps, sont parvenus à stabiliser le centre Fe-S de la RirA et à modéliser partiellement la structure de cette protéine. Ceci a permis à un peu mieux comprendre comment cette protéine régule les niveaux de fer intracellulaires. Gray E et al., « Stabilisation of the RirA [4Fe-4S] cluster results in loss of iron-sensing function ». Chemical Science, 2023 August 22;14(36):9744-9758.
[4] Volbeda A. et al., « Crystal Structure of the Transcription Regulator RsrR Reveals a [2Fe-2S] Cluster Coordinated by Cys, Glu, and His Residues », Journal of the American Chemical Society, 2019 February 13;141(6):2367-2375.
[5] Crack J.C. et al., « Electron and proton transfers modulate DNA binding by the transcription regulator RsrR ». Journal of the American Chemical Society, 2020, February 20; 142:5104-5116.
[6] Rohac R. et al., « Structural determinants of DNA recognition by the NO sensor NsrR and related Rrf2-type [FeS]-transcription factors », Communications Biology, 2022 July 30;5(1):769.



