Vous êtes ici
Sur la piste des génomes artificiels

Votre domaine de recherche est au croisement de l’informatique et de la génétique. Quelles sont les grandes questions que vous vous posez ?
Flora Jay1. Pour préciser, je dirai que mon champ de recherche est entre l'informatique, la génétique, mais aussi les mathématiques et la statistique. À partir de données génétiques, j’essaie de reconstruire l'histoire démographique des populations. Par exemple, comment des espèces ou des populations se sont séparées puis de nouveau mélangées, ou comment certains facteurs ou événements ont conduit à la sélection de certaines versions des gènes. Dans mes recherches, il y a des aspects écologiques, historiques, médicaux.
Vous avez publié cette année une recherche (en anglais) qui a fait sensation dans votre communauté : la création, grâce à des réseaux de neurones artificiels, des morceaux génomes réalistes. Pourriez-vous nous rappeler tout d’abord ce que sont les neurones artificiels ?
F. J. Un réseau de neurones est un modèle statistique qui permet d’apprendre et d’approcher des fonctions complexes. En l’entraînant sur un jeu de données, il peut apprendre à réaliser des tâches difficiles.
Par exemple, supposons que vous voulez l’entraîner à reconnaître un chat d’un chien sur des images. Au début, le réseau choisit au hasard et se trompe une fois sur deux. Mais, à force de se tromper et de se corriger, il finit par apprendre à les distinguer. Avec Burak Yelmen, premier auteur du papier, nous nous sommes demandé si ces méthodes de machine-learning (apprentissage machine) pourraient nous aider à imiter des données génétiques.
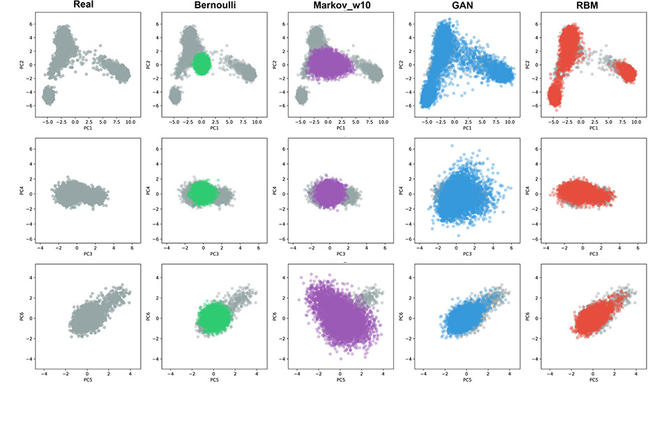
Nous sommes parvenus à entraîner un réseau de neurones sur une grande base de génomes, la biobanque estonienne, et une fois entraîné, nous avons pu créer des portions de génomes réalistes. Ces bouts de génomes gardent les traits caractéristiques des génomes d’origine, mais en réalité, ils n'appartiennent à aucun individu véritable.
Vous obtenez une sorte de portrait-robot des génomes de la base de données ?
F. J. Pas tout à fait. Un portrait-robot cherche à imiter le visage de quelqu’un. Ici, on n’imite pas un génome en particulier. On obtient des séquences réalistes, impossibles à distinguer d’un vrai génome, mais qui n’appartiennent à personne. C’est plutôt comme ces visages synthétiques, créés par ordinateur, qui n’existent pas dans la réalité.
Quelles sont les applications possibles de ces génomes artificiels ?
F. J. De nombreuses banques génétiques, en particulier privées, ne permettent pas l’accès à leurs données aux chercheurs pour des raisons financières, mais parfois aussi de confidentialité.
En effet, lorsqu’on a beaucoup de génomes et si la population d’où ils sont issus n’est pas très grande, on peut finir par reconnaître à qui ils appartiennent à partir des liens familiaux et d’informations comme l’âge ou des traits caractéristiques. Cela pose des problèmes éthiques car, à partir de génomes on peut remonter de manière plus ou moins fiable, à des informations confidentielles comme l’ascendance génétique, des liens de parentalité, des risques augmentés d’avoir une maladie. Or, les données publiques ont permis de nombreuses découvertes en génétique des populations et il serait heureux que les chercheurs puissent aussi utiliser les données privées.
Notre méthode pourrait apporter une solution. Grâce à l’apprentissage machine, on pourrait construire des génomes artificiels qui procurent des informations importantes sur la population étudiée, mais qui n’appartiennent à aucun individu véritable. Ces génomes artificiels pourraient alors servir à la recherche biomédicale ou génétique des populations, ou autres.
De quelle manière ?
F. J. Prenons par exemple un chercheur qui s’intéresse à la part de génome de Néandertal qu’il y a dans une population contemporaine. Si, pour des raisons de confidentialité, il n’a pas accès aux données génétiques réelles de cette population, il pourrait utiliser comme proxy nos génomes artificiels. Créés à partir des données génétiques de cette population, ils en garderaient les caractéristiques, et devraient donc aussi garder la trace de cet ADN ancien.
Y a-t-il une autre application à ces méthodes ?
F. J. C’est encore très prospectif, mais grâce à l’entraînement de réseaux de neurones, on peut identifier des régions atypiques sur lesquelles s’est peut-être exercée une pression de sélection. Par exemple, la mutation qui permet la digestion du lactose pourrait ainsi ressortir, ou alors des régions liées à la résistance à des pathogènes. Une fois identifiées, les chercheurs pourraient se pencher plus en détail sur ces régions.
Pouvez-vous me décrire votre parcours, et ce qui vous a menée vers ces thématiques ?
F. J. J’ai fait une école d'ingénieur avec spécialités mathématiques appliquées et informatique, et un master en informatique et mathématiques appliquées à la biologie. J'ai ensuite fait une thèse de modélisation et de statistiques pour la biologie des populations, puis un postdoctorat à Berkeley en 2012. Là, j’ai travaillé sur l’ADN de deux femmes, néandertalienne et dénisovienne. On voulait comprendre de quelle manière, quand, et en quelle proportion les Néandertaliens, Dénisoviens et Homo sapiens s’étaient mélangés. Je suis ensuite revenue en France et j'ai travaillé à des reconstructions d'histoire démographique et au développement de méthodes statistiques pour résumer les génomes sans perdre trop d’information. Je me suis alors demandé si on pouvait construire des architectures neuronales adaptées pour extraire l'information des génomes de manière plus automatique. C'est comme ça que je suis arrivée au Laboratoire interdisciplinaire des sciences du numérique fin 2015.
Qu’est-ce qui vous motive le plus dans votre travail ? Est-ce que ce sont les questions sur la génétique et l'histoire évolutive, ou plutôt le développement de nouvelles méthodes d'analyse ?
F. J. C'est difficile de répondre à cette question. J'aime travailler en collaboration parce que, quand on travaille avec différents chercheurs, le rôle de chacun change. On peut être la personne qui se penche sur la méthodologie pour répondre à la question de notre collaborateur, ou bien, on peut être la personne qui emmène la question biologique. Mais c'est vrai que j'aime bien développer des outils qui ensuite seront disponibles à tout le monde.
Ces collaborations ne doivent pas toujours être faciles. Vous travaillez par exemple avec des biologistes qui ne parlent pas le même langage que vous.
F. J. Cela demande du temps des deux côtés, mais c'est enrichissant. Parfois, c'est de l'incompréhension que naissent des projets intéressants. Parfois, quand on ne se comprend pas, on pense soudain à quelque chose que ni l’un ni l’autre n'avait pas envisagé. Quelque chose d’hybride peut émerger.
Quelle suite allez-vous donner à ce travail ?
F. J. Burak Yelmen vient de rejoindre le laboratoire et, avec l’équipe Tau d’Inria, nous avons plein d’idées. Par exemple, nous voulons trouver une méthode élégante afin de passer de portions de génomes de quelques millions de paires de bases à un génome artificiel complet qui en contiendrait environ trois milliards. Nous voulons aussi développer des indicateurs qui permettent d’évaluer le degré de réalisme des génomes artificiels et de nous assurer qu’il n’y a pas de fuite d’information à partir de génomes réels. Un autre volet de mes travaux avec les réseaux de neurones est de les utiliser dans la recherche sur les bactéries, en collaboration avec Jean Cury. Le but est d’utiliser ces méthodes d’apprentissage statistique pour suivre l’évolution de populations bactériennes et voir l’impact d’événements comme l’arrivée de nouveaux antibiotiques. ♦
Référence
"Creating artificial human genomes using generative neural networks", B. Yelmen, A. Decelle, L. Ongaro et al., 2021, PLoS Genet 17(2): e1009303. . https:// doi.org/10.1371/journal.pgen.1009303
- 1. Laboratoire interdisciplinaire des sciences du numérique (CNRS/Université Paris-Saclay).