A la une
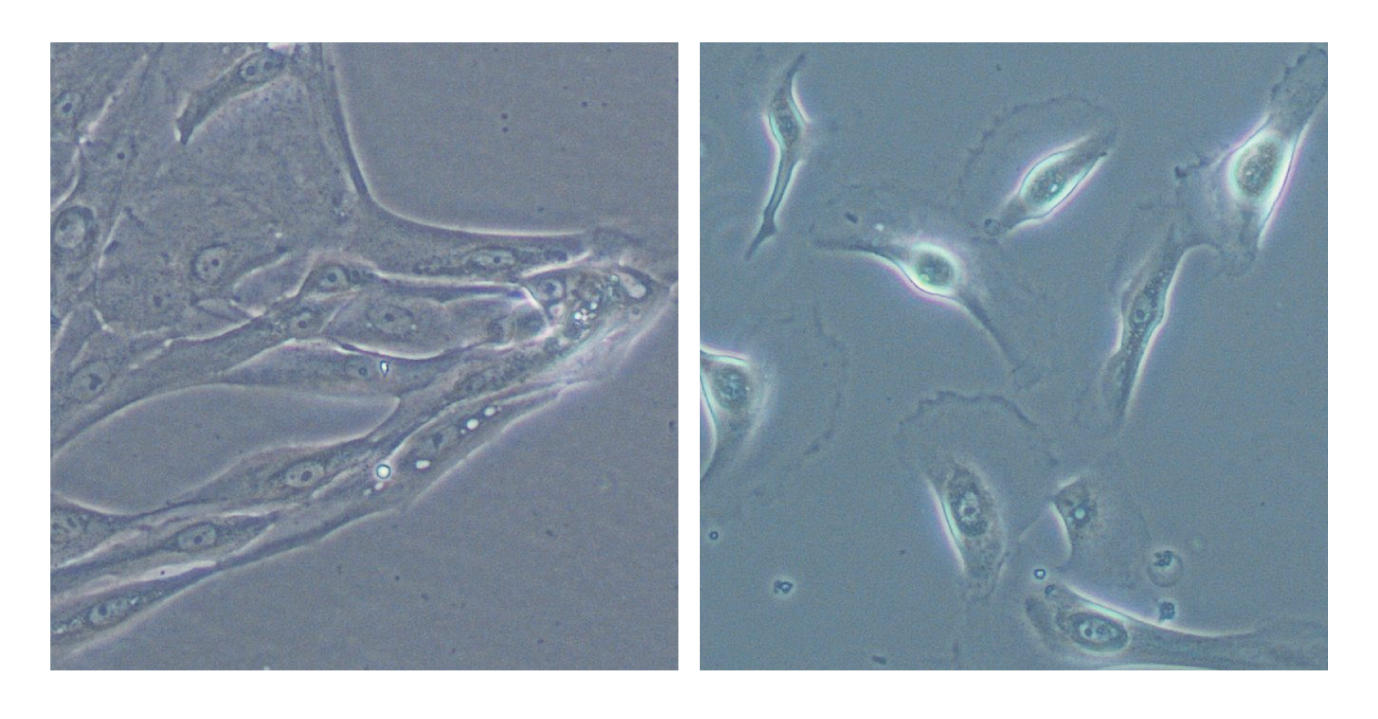
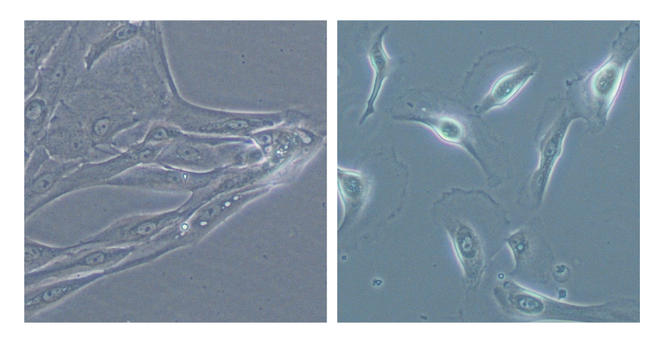
Toutes sortes de cellules peuvent devenir cancéreuses, et elles présentent un niveau de menace différent. À la fois résistantes, actives et mobiles, les cellules souches cancéreuses font croître les tumeurs et provoquent des récidives de cancer, même après traitement. Elles forment ainsi des cibles importantes pour la recherche.
Des molécules pour capturer le fer
« Ces cellules cancéreuses résistantes contiennent plus de fer et sont métaboliquement plus actives que les autres, explique Raphaël Rodriguez, directeur de recherche CNRS au laboratoire Chimie et biologie de la cellule1. Nous avons donc décidé de les attaquer par cet angle. » Raphaël Rodriguez a choisi l’ironomycine, petite molécule de synthèse dérivée de la salinomycine conçue dans son laboratoire, qu’il a brevetée. Le produit naturel ainsi que ce dérivé de synthèse bloquent le fer lysosomal et perturbent l’homéostasie de la cellule. L’accumulation de ce métal entraîne la production d’espèce réactives de l’oxygène toxiques pour la tumeur. Cela finit par enclencher le processus de mort cellulaire des cellules souches cancéreuses.
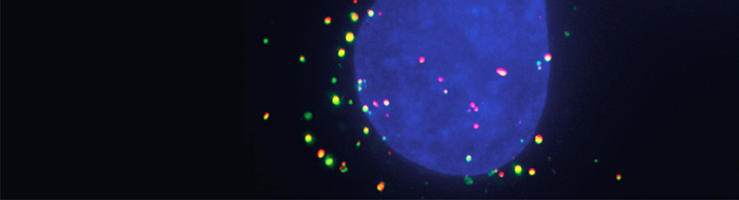
Forts de cette piste extrêmement prometteuse et inédite, Raphaël Rodriguez et Robin Rivaton, économiste et essayiste, ont fondé une start-up pour valoriser l’ironomycine. « Il a d’abord fallu publier beaucoup de travail académique, puis discuter avec des contacts dans les biotechnologies et les milieux économiques, se souvient le chimiste. Lancer une start-up demande le bon timing. Ni trop tôt ni trop tard. » Une approche d’influence anglo-saxonne, inspirée par son année de thèse à Oxford et ses sept années de postdoc à Cambridge.
SideROS naît alors en 2019, soutenue par le programme RISE de CNRS Innovation. Sideros signifie « fer » en grec, mais les trois dernières lettres sont ici capitalisées pour ROS : reactive oxygen species. Un terme qui désigne les dérivés réactifs de l’oxygène, comme les radicaux libres, formés en perturbant l’homéostasie du fer dans les cellules souches cancéreuses.
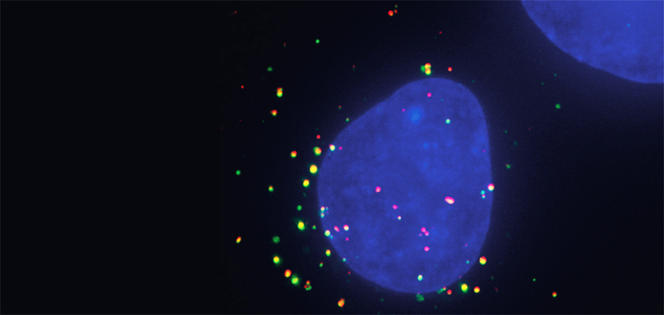
Reconnaissance académique et attractivité
La jeune pousse est en tout cas immédiatement repérée, et reçoit la même année le prix de la start-up innovante du prestigieux concours i-LAB, organisé par le ministère de l’Enseignement supérieur, de la Recherche et de l’Innovation, en partenariat avec la Banque publique d’investissement (BpiFrance). Toujours en 2019, Raphaël Rodriguez obtient non seulement le grand prix scientifique de la Fondation Charles Defforey, remis à l’Institut de France, mais aussi le Tetrahedron Young Investigator Award pour l'ensemble de ses travaux, prix international réservé aux chercheurs de moins de quarante ans. « Le concours i-Lab nous a offert une visibilité très importante, s’enthousiasme Lucie Mondoulet, Chief executive officer (CEO) de SideROS. Notre dossier a ainsi été reconnu par des experts scientifiques de très haut niveau, ce qui renforce la solidité de notre dossier pour la recherche de financement. »
Ancienne chercheuse en génie biochimique et alimentaire, spécialisée dans la désensibilisation aux allergies alimentaires, Lucie Mondoulet a créé en 2007 le département Recherche et Développement de DBV Technologies. Pendant les onze années où Lucie Mondoulet a dirigé ce département, la petite entreprise française d’une dizaine de personnes est passée à trois cents salariés, a été valorisée jusqu’à deux milliards d’euros et est toujours cotée au Nasdaq et à la Bourse de Paris, devenue Euronext.
« J’ai quitté DBV en 2018, pour reprendre un projet de recherche dès ses premières phases de découverte, avant toutes les étapes de développement nécessaires pour démarrer les essais cliniques, explique Lucie Mondoulet. C’est la période la plus critique, mais c’est aussi là où l’on retrouve le plus d’enjeux et de créativité, pour adapter ce projet de recherche en projet de développement de médicaments, ainsi que convaincre les investisseurs et partenaires industriels pour les premières étapes de financement de la société. »
Vers des essais cliniques sur les humains
Un ancien collègue de DBV l’avait mise en contact avec Raphaël Rodriguez, qui cherchait une personne expérimentée pour planifier comment amener ses molécules jusqu’aux essais cliniques. D’abord consultante, Lucie Mondoulet est devenue CEO de SideROS en septembre 2019. Pour les prochains mois, le travail consistera à industrialiser les procédés de fabrication maîtrisés à l’échelle du laboratoire et évaluer la toxicité de l’ironomycine avant une première administration chez le patient.
« Nos tests in vitro ont confirmé l’efficacité de la molécule sur des cellules de cancers hématologiques2, ainsi que de cancers solides tels que le glioblastome3 ou celui des ovaires, détaille Lucie Mondoulet. Nous avons aussi des preuves de concept précliniques in vivo sur des souris greffées avec une tumeur du sein, qui montrent qu’elle empêche la reprise tumorale. Nous espérons que les premiers essais cliniques sur les humains pourront commencer en 2022, qu’il faudra alors financer par une levée de fonds à hauteur de plusieurs dizaines de millions d’euros. » Le défi est ainsi triple : scientifique, financier et sanitaire. L’ironomycine recèle cependant bien assez de promesses pour que l’équipe de SideROS les relève tous. ♦