A la une
Après dix années de travaux, un consortium de chercheurs est en passe de parfaire la compréhension des cibles thérapeutiques, ouvrant la voie à l’amélioration de nombreux traitements. Explications dans ce nouveau billet du blog « Focus Sciences ».
Près de 70% des médicaments, qu’ils soient administrés pour traiter une légère infection ou un cancer, ciblent des protéines localisées dans la membrane cellulaire, la zone qui entoure chaque cellule. Il est essentiel de parvenir à bien décrypter et décrire ces protéines membranaires (MPs) pour concevoir ensuite des médicaments qui les atteignent efficacement. Toutefois, cette étape a donné du fil à retordre à des générations de scientifiques, puisqu’elle nécessite d’extraire et d’isoler ces protéines à l’aide de détergents qui peuvent parfois dégrader leur structure.
Des détergents qui brouillent l’analyse des cibles thérapeutiques
« Nous étions confrontés au fait que ces protéines devenaient instables dès lors qu’elles sortaient de leur contexte membranaire », précise Pierre Falson, directeur de recherche CNRS au laboratoire microbiologie moléculaire et biochimie structurale1. Dès lors, certaines de leurs fonctions biochimiques observées en laboratoire étaient altérées et différentes de celles qui se produisent naturellement dans la membrane plasmique. Les données permettant d’améliorer la liaison des médicaments aux récepteurs qu’ils ciblent peuvent alors manquer de précision et, finalement, nuire à l’élaboration d’un médicament efficace.
Depuis près de dix ans, Pierre Falson a pris les rênes d’un groupement international de chimistes, biochimistes et biologistes pour tenter de résoudre ce problème grâce à la création et l'évaluation de détergents plus « doux ». L’objectif étant de former un milieu stabilisateur qui maintient et préserve la structure des MPs lors de leur analyse. Une fois les protéines intactes isolées, les scientifiques passent à l’étape de la cristallographie pour étudier précisément leur structure tridimensionnelle. Cette technique d’imagerie repose sur la capacité des molécules à se « cristalliser », c'est-à-dire à s'organiser de manière régulière dans l'espace pour former des cristaux. Les chercheurs utilisent alors la diffraction (déviation de rayons) obtenue par le « bombardement » du cristal avec des faisceaux de particules pour déterminer la position exacte (et en 3D) des atomes qui composent les protéines membranaires.
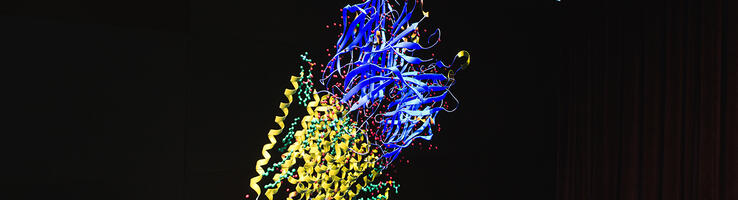
 Visualisation de la structure d’une protéine membranaire de type canal ionique © Frédérique Plas / IBPC / CNRS Images
Visualisation de la structure d’une protéine membranaire de type canal ionique © Frédérique Plas / IBPC / CNRS Images
Une précision jamais atteinte dans la description des protéines membranaires
Ces recherches peuvent être comparées à l'évolution de la photographie pour mieux comprendre les avancées qu'elles ont permises. Les détergents classiques, similaires aux premières solutions de traitement et de développement photographiques, altèrent suffisamment les protéines membranaires pour que la structure de ces dernières soit peu précise. L'objectif était donc de développer une solution plus douce pour révéler la structure de la protéine dans ses moindres détails, tout comme les procédés optiques les plus récents permettent de révéler des images de haute résolution, allant jusqu’au GigaPixel, soit un milliard de pixels. Pierre Falson préfère filer la métaphore serrurière : « si le médicament est une clé, alors la serrure sera la structure moléculaire de la cible, ici les MPs. Notre recherche s’attache donc à mieux connaître ces serrures pour produire des clés sans défectuosité ».
Grâce à leurs travaux sans précédents, les chercheurs ont réussi à concevoir deux concepts de détergents qui ont été brevetés, parmi lesquels les DCODs (glycosyl-substituted dicarboxylate detergents) dont la structure chimique modifiée a considérablement amélioré l'extraction des MPs. L'efficacité du caractère « stabilisant » de ces détergents est mesurée en termes de décalage thermique, c'est-à-dire en comparant les températures auxquelles les MPs se dégradent en présence et en l'absence de détergent. Un décalage thermique considéré comme « positif » se produit lorsque la température à laquelle la protéine se dégrade est plus élevée en présence de détergent qu'en son absence ; on considère alors que son milieu est plus protecteur. Dans le cadre des expériences sur les DCODs, la protéine BmrA, qui aide la bactérie Bacillus subtilis à contrôler la résistance aux antibiotiques, a ainsi toléré des températures plus élevées de 30°C grâce à ces stabilisants.
Un procédé nobélisé dans l’équation
Dans une nouvelle étape, les chercheurs tentent d'améliorer encore ce procédé d’analyse des protéines, grâce à une autre technique d’imagerie apportée par l’équipe suédoise associée au projet : la microscopie cryo-électronique.
Nobélisé en 2017, ce procédé utilise des échantillons cryogénisés (préservés à froid dans leur état natif) et permet de visualiser des structures complexes à des résolutions très élevées, allant jusqu'à 2-3 Ångströms (1 Ångström = 1 dixième de nanomètre, soit un dix-milliardième de mètre, l’échelle de taille d’un atome). Grâce à cette précision, les chercheurs peuvent déterminer la localisation exacte des domaines actifs et des sites d'interaction des protéines avec d'autres molécules ; une information cruciale pour identifier les sites cibles des médicaments.
Les chercheurs ont ainsi pu comprendre les mécanismes, jusqu’alors inconnus, par lesquels les transporteurs ABC (protéines transmembranaires) permettent à certaines substances médicamenteuses de franchir la membrane plasmique. Une découverte majeure, qui a fait l’objet d’un article publié en 2022 dans le journal Science Advances.
Une innovation peut en cacher une autre
Pour Ahcène Boumendjel, Professeur des Universités rattaché au Laboratoire des radiopharmaceutiques biocliniques2, le succès et la rapidité d’innovation de ce projet résident dans « la combinaison fructueuse des expertises en chimie médicinale, biochimie et biologie structurale ». Cette multidisciplinarité offre deux promesses pour le développement de nouveaux médicaments : tout d'abord, les chercheurs peuvent espérer concevoir des molécules qui se lient spécifiquement aux sites cibles identifiés, ce qui permet d'éviter des effets secondaires indésirables. Ensuite, le processus de développement des médicaments (habituellement long d'une quinzaine d'années) pourrait être considérablement réduit en disposant très tôt de données précises sur la structure des protéines cibles des futurs candidats médicaments.
Quant à savoir s’il n’est pas plutôt possible de se passer de détergents pour isoler ces protéines, « c’est envisageable, oui, mais pas tout de suite, loin de là », répond Pierre Falson. Il anticipe d’ailleurs une véritable révolution de la biologie structurale dans les prochaines décennies avec le développement d’AlphaFold. Un outil basé sur de l’intelligence artificielle potentiellement capable de prédire les structures des protéines, mais qui est encore loin d’être pleinement efficace. D’ici là, les solutions apportées par ces chercheurs permettront à la communauté scientifique de parfaire sa compréhension des protéines membranaires et de lutter ainsi plus efficacement contre de nombreuses maladies.
Cet article fait partie d’un dossier thématique sur l’antibiorésistance "Résistances aux traitements : la recherche en quête de solutions" réalisé en collaboration avec Pop’Sciences - Université de Lyon (mai 2023).
--------------------
Ces recherches ont été financées en tout ou partie par l'Agence Nationale de la Recherche (ANR) au titre du projet ANR Clamp2-AAPG2018. Cette communication est réalisée et financée dans le cadre de l'appel à projet Sciences Avec et Pour la Société - Culture Scientifique Technique et Industrielle pour les projets JCJC et PRC des appels à projets génériques 2018-2019 (SAPS-CSTI-JCJC et PRC AAPG 18/19).



