Vous êtes ici
Diagnostiquer à l’échelle nanométrique
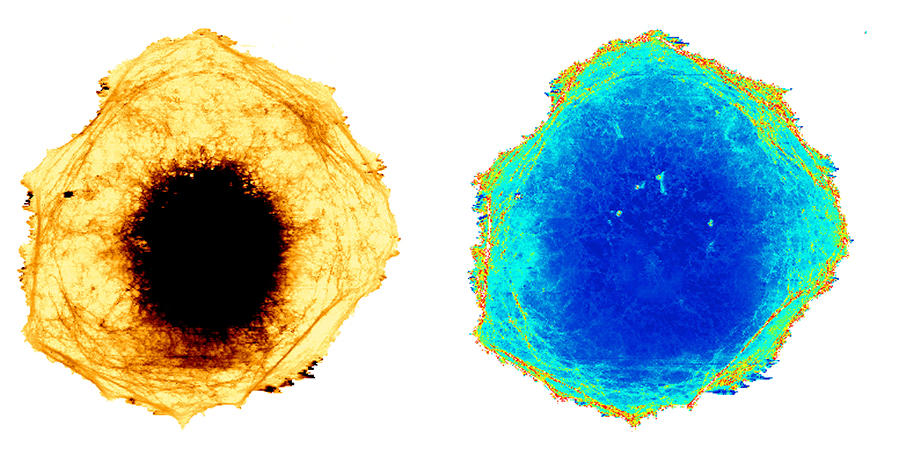
Votre équipe de recherche CNRS de l’Institut Pasteur de Lille a réussi1 à combiner trois modes d’imagerie dans une approche dite « corrélative », c’est-à-dire, sur le même échantillon. Quelles opportunités offre cette technique innovante ?
Frank Lafont : Nous pouvons enfin voir et palper le vivant à l’échelle nanométrique ! Cette approche associe trois techniques : la microscopie de champ proche, qui permet d’imager des objets à partir du suivi de la déflexion d’une pointe nanométrique, la microscopie photonique de superrésolution, qui utilise le suivi de molécules fluorescentes, la microscopie électronique, qui permet d’obtenir des informations sur des ultrastructures cellulaires denses aux électrons. Nous sommes ainsi capables aujourd’hui d’observer des cellules et des organites intracellulaires en activité avec une précision inégalée, d’analyser leurs propriétés biophysiques (mesures d’adhérence, forces d’interactions, élasticité et viscosité) et de corréler ces données au niveau ultrastructural dans la cellule.
Quels sont les champs d’application possibles ?
F.L. : Les applications sont multiples, en particulier en médecine et dans la santé, et en cosmétologie. Nous pouvons mesurer les paramètres biophysiques d’un échantillon vivant sur une échelle de temps donnée et donc suivre leur évolution dans des situations physiopathologiques. En analysant l’élasticité des cellules tumorales à différents stades de développement, il devient par exemple possible de définir une échelle de référence des signes d’occurrence d’une tumeur. L’analyse fine des transformations physiques du vivant constitue de fait une aide unique au diagnostic. Mais ce n’est pas tout. Cette technique permet par exemple de suivre les perturbations membranaires des cellules de la peau liées à l’âge ou au développement du diabète. Ou encore les altérations mécaniques des cellules, lors de processus infectieux ou neurodégénératifs. Outre cette aide au diagnostic, notre approche corrélative offre la possibilité de tester l’efficacité de multiples molécules, développées en pharmacologie ou en cosmétologie pour corriger ces dysfonctionnements. Des partenariats sont d’ailleurs en cours avec des industriels notamment sur les différentes applications de la microscopie à force atomique, ou AFMFermerPour Atomic Force Microscope., que nous développons pour le domaine biomédical et qui peut aussi être utilisée pour la nanocaractérisation de matériels mous en liquide pour l’industrie des matériaux.
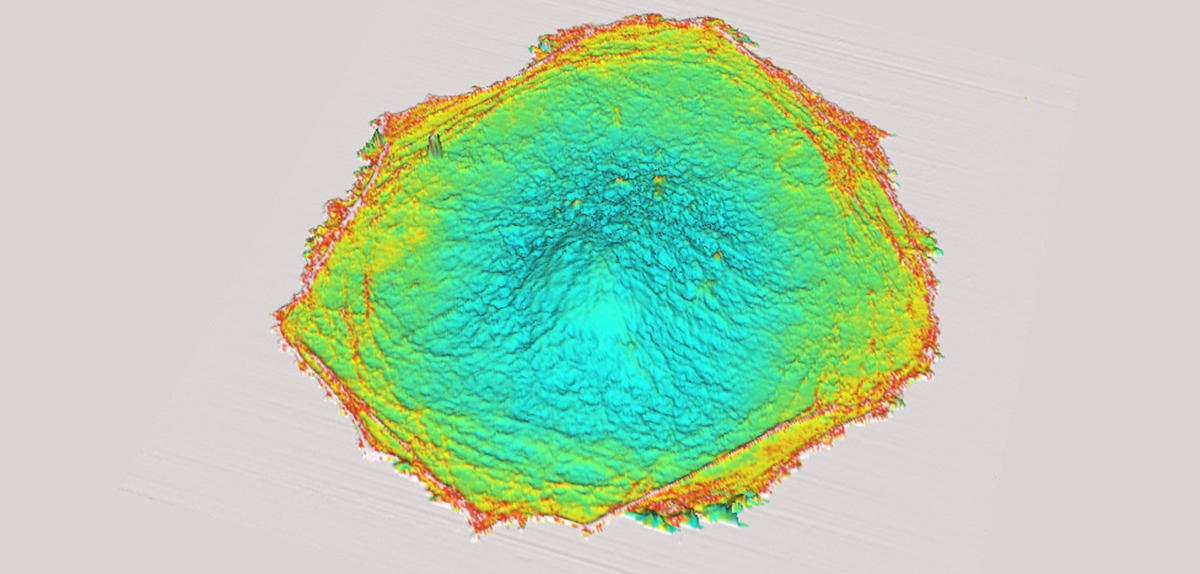
Quel est le principe de cette technique ?
F.L. : L’AFM dérive de la microscopie à effet tunnel et permet d’effectuer des mesures biophysiques sur un échantillon vivant avec une précision nanométrique. Le principe consiste à approcher une pointe très fine à proximité immédiate, voire au contact de l’échantillon. Comme celle-ci repose sur un microlevier flexible, dont le déplacement est suivi par un faisceau laser, son utilisateur peut « palper » l’échantillon, au sens littéral du terme, tel un doigt qui discerne les parties molles (tissus) et les parties dures (os, tendons) d’une main, et d’en révéler simultanément les caractéristiques mécanobiologiques.
On peut ainsi mesurer l’influence du squelette cellulaire sur la formation de tunnels transcellulaires provoqué par une toxine du staphylocoque doré sur une cellule endothélialeFermerEn contact direct avec le sang, les cellules endothéliales tapissent l'intérieur des vaisseaux. ou sur les interactions entre une molécule de surface du méningocoque fixée sur la pointe et son récepteur sur cette même cellule.
Sur quelles innovations techniques récentes vous êtes-vous appuyés ?
F.L. : Notre approche s’appuie sur les innovations majeures qui ont émergé ces dernières années.
Tout d’abord, la méthode ClemFermerPour Correlative Light Electron Microscopy. qui permet de combiner des informations sur la structure globale des cellules, obtenues en microscopie électronique, avec l’analyse de la dynamique fonctionnelle de leurs organites en microscopie photonique. Une caractéristique commune à de nombreuses maladies neurodégénératives, dont la maladie de Huntington et la maladie d’Alzheimer, est l’accumulation de protéines mutées ou toxiques qui s’agrègent dans la cellule. Elles sont les témoins du dysfonctionnement du mécanisme de nettoyage et de recyclages des déchets cellulaires (autophagie). Nous appliquons d’ailleurs la Clem pour caractériser les vacuolesFermerPrésentes dans les cellules des végétaux, les vacuoles sont de petites structures, appelées organites, qui jouent un rôle protecteur et interviennent dans les mécanismes de défense. De nombreuses avancées ont pu être réalisées dans ce domaine depuis le milieu des années 1990, précisément grâce à la méthode Clem. Ensuite, l’avènement des techniques photoniques à résolution nanométrique, dites de « superrésolution », a donné naissance à la super-Clem. Et notre équipe de recherche a hissé d’un cran cette super-Clem en y intégrant l’AFM.
Plus largement, quelles sont les recherches menées en Europe et à l’international ?
F.L. : L’espace européen de la recherche est l’une des têtes de pont de l’innovation à l’échelle internationale. Plusieurs équipes de recherche de taille restreinte mais très performantes sont à pied d’œuvre à travers le monde. En France, nous collaborons étroitement avec deux centres de recherche majeurs qui utilisent les dernières techniques de l’AFM à haute vitesse développée au début des années 2000 pour étudier les processus moléculaires rapides in vivo (HS-AFM). Les travaux de l’équipe de Pierre-Emmanuel Milhiet, du Centre de biochimie des membranes de Montpellier,2 ont permis d’étudier la formation de structures fibrillaires de type amyloïde ou, plus classiquement, d’analyser des étapes de fusion du virus de la grippe. Avec la même approche, Félix Rico et ses collaborateurs, du laboratoire Adhésion et inflammation (LAI)3 concentrent leurs efforts sur les réponses viscoélastiques des cellules. Nous pensons que la corrélation entre superrésolution, HS-AFM permettra bientôt de voir les réactions des machines moléculaires en direct sur les cellules. Nous avons monté avec ces équipes, des collègues de Grenoble et une dizaine de laboratoires européens, un consortium européen au sein du programme EU-COST AFM4NanoMed&Bio pour établir une procédure standard et favoriser enfin les analyses comparatives des résultats de recherche en élasticité cellulaire reposant sur l’AFM.
La suite de cette collaboration européenne pourrait permettre des applications comme le suivi direct sur organe ou sur biopsies de l’élasticité des tissus en fonction de troubles physiopathologiques. Notre apport dans ce contexte est d’avoir pu établir une corrélation entre des méthodes dont l’intérêt a été reconnu par plusieurs prix Nobel : microscopie électronique et microscopie à effet tunnel en 1986 (à Ernst Ruska, Gerd Binnig et Heinrich Rohrer), l’utilisation de sondes fluorescentes en 2008 (à Osamu Shimomura, Martin Chalfie et Roger Tsien) et la superrésolution en 2014 (à Stefan Hell et William Moerner). Enfin, je salue bien évidemment les travaux du Suisse Jacques Dubochet, de l'Allemand Joachim Frank et de l’Écossais Richard Henderson, pour le développement de la cryo-microscopie électronique (cryo-EM)FermerCette technique de microscopie électronique utilise le froid lors de l’étape de fixation (cryo signifie « froid » en grec ancien). Il n’est donc pas nécessaire de colorer l’échantillon. qui ont été récompensés par le prix Nobel de chimie fin 2017. La cryo-EM a déjà fourni des images inédites en 3D des protéines responsables de la résistance aux antibiotiques et de la surface du virus Zika. On perçoit bien que l’imagerie cellulaire et tissulaire a encore beaucoup à nous apprendre dans ses développements pour comprendre les réactions moléculaires in cellulo en 3D et en fonction de la situation physique de la cellule (étirée, comprimée, en déplacement, en interaction avec une autre cellule ou un agent pathogène…). ♦
- 1. « CLAFEM: Correlative light atomic force electron microscopy », S. Janel, E. Werkmeister, A. Bongiovanni, F. Lafont, N. Barois, « Methods Cell Biol. », 2017;140:165-185. doi: 10.1016/bs.mcb.2017.03.010. Epub 2017 Apr 21.
- 2. Unité CNRS/Inserm/Université de Montpellier.
- 3. Unité CNRS/Inserm/ Université Aix Marseille
Voir aussi
Auteur
Séverine Lemaire-Duparcq est journaliste scientifique.