A la une

Dans les années 1950, le cycle de Calvin est mis à jour. Pendant la photosynthèse1, cette suite de réactions biochimiques permet aux plantes de fixer le carbone et de le transformer notamment en glucose. Depuis, les grandes phases chimiques ont été largement étudiées, mais les protéines métaboliques impliquées, elles, sont restées en retrait. C’est là qu’intervient le projet ANR CALVINTERACT, porté par Julien Henri, enseignant-chercheur au laboratoire Biologie moléculaire et cellulaire des eucaryotes, puis au laboratoire Biologie computationnelle quantitative et synthétique : de la cellule à la structure moléculaire 3D2. Il s’est lancé dans un chantier ambitieux – isoler, produire et cartographier l’ensemble des enzymes3 du cycle chez des micro-algues. Une véritable boucle de recherche, répétée, modélisée et affinée.
Photos : Christophe Hargoues
 Première pièce du puzzle. Modéliser la structure d’une protéine consiste à identifier le positionnement de chaque atome dans la molécule. Pour aboutir à une représentation numérique comme ici, il faut d’abord passer par de nombreuses étapes à la paillasse.
Première pièce du puzzle. Modéliser la structure d’une protéine consiste à identifier le positionnement de chaque atome dans la molécule. Pour aboutir à une représentation numérique comme ici, il faut d’abord passer par de nombreuses étapes à la paillasse.
 Sélection des micro-algues. Étalement sur boîte des colonies de Chlamydomonas reinhardtii, une micro-algue modèle utilisée dans l’étude de la photosynthèse. Cette étape de sélection permet d’identifier les lignées viables, stables et reproductibles, capables de produire suffisamment d’ARN messager4 pour isoler les gènes d’intérêt : ceux qui codent la production des protéines du cycle de Calvin.
Sélection des micro-algues. Étalement sur boîte des colonies de Chlamydomonas reinhardtii, une micro-algue modèle utilisée dans l’étude de la photosynthèse. Cette étape de sélection permet d’identifier les lignées viables, stables et reproductibles, capables de produire suffisamment d’ARN messager4 pour isoler les gènes d’intérêt : ceux qui codent la production des protéines du cycle de Calvin.
 Culture liquide des lignées sélectionnées. Les lignées retenues sont cultivées dans un milieu liquide sous lumière contrôlée. Cette phase de croissance permet d’obtenir suffisamment de biomasse – matière vivante issue de la culture (les cellules cultivées) – pour extraire les gènes codant les enzymes du cycle de Calvin.
Culture liquide des lignées sélectionnées. Les lignées retenues sont cultivées dans un milieu liquide sous lumière contrôlée. Cette phase de croissance permet d’obtenir suffisamment de biomasse – matière vivante issue de la culture (les cellules cultivées) – pour extraire les gènes codant les enzymes du cycle de Calvin.
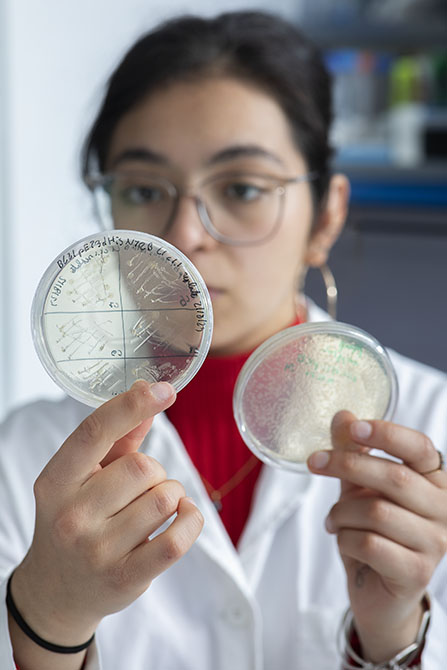 Colonies bactériennes sur boîtes. La micro-algue produit naturellement ces enzymes, mais en trop faible quantité, et noyées dans un foisonnement d’autres protéines végétales. Pour isoler chacune d’elles, les chercheurs extraient son gène et le transfèrent dans des bactéries, modifiées pour produire une seule protéine. Dans des boîtes de culture, ils identifient les souches capables d’exprimer efficacement cette dernière.
Colonies bactériennes sur boîtes. La micro-algue produit naturellement ces enzymes, mais en trop faible quantité, et noyées dans un foisonnement d’autres protéines végétales. Pour isoler chacune d’elles, les chercheurs extraient son gène et le transfèrent dans des bactéries, modifiées pour produire une seule protéine. Dans des boîtes de culture, ils identifient les souches capables d’exprimer efficacement cette dernière.
 Culture liquide des bactéries. Les bactéries sélectionnées deviennent de véritables mini-usines à enzymes. Cultivées en fioles dans des milieux liquides, elles produisent des quantités suffisantes de protéines, prêtes à être extraites et purifiées. Une stratégie de production dite hétérologue, simple mais redoutablement efficace.
Culture liquide des bactéries. Les bactéries sélectionnées deviennent de véritables mini-usines à enzymes. Cultivées en fioles dans des milieux liquides, elles produisent des quantités suffisantes de protéines, prêtes à être extraites et purifiées. Une stratégie de production dite hétérologue, simple mais redoutablement efficace.
 Démarrage de la purification. Place à la purification : après le broyage des bactéries, les protéines sont extraites puis séparées selon leurs propriétés. Au premier plan, une colonne de chromatographie d’affinité pour l’isolement de l’enzyme cible. À l’arrière, une étape ultérieure permet de poursuivre l’enrichissement sélectif en protéine cible.
Démarrage de la purification. Place à la purification : après le broyage des bactéries, les protéines sont extraites puis séparées selon leurs propriétés. Au premier plan, une colonne de chromatographie d’affinité pour l’isolement de l’enzyme cible. À l’arrière, une étape ultérieure permet de poursuivre l’enrichissement sélectif en protéine cible.
 Contrôle qualité par électrophorèse. Sur ce gel, chaque bande bleue correspond à une protéine. Cette méthode, dite d’électrophorèse, permet de vérifier la pureté de l’échantillon. Ici, la piste centrale avec une bande unique indique que la protéine isolée est pure, un prérequis à la cristallisation.
Contrôle qualité par électrophorèse. Sur ce gel, chaque bande bleue correspond à une protéine. Cette méthode, dite d’électrophorèse, permet de vérifier la pureté de l’échantillon. Ici, la piste centrale avec une bande unique indique que la protéine isolée est pure, un prérequis à la cristallisation.
 Concentration des échantillons. La protéine purifiée est transférée dans des tubes pour augmenter sa concentration en réduisant le volume de l’échantillon. Cette étape est indispensable pour favoriser ensuite la cristallisation qui nécessite que les molécules soient suffisamment proches pour s’ordonner en réseau.
Concentration des échantillons. La protéine purifiée est transférée dans des tubes pour augmenter sa concentration en réduisant le volume de l’échantillon. Cette étape est indispensable pour favoriser ensuite la cristallisation qui nécessite que les molécules soient suffisamment proches pour s’ordonner en réseau.
 Quantification des protéines par spectrophotométrie. La spectrophotométrie permet de détecter et de quantifier les protéines concentrées. C’est une étape de validation cruciale avant l’analyse structurale : si les conditions sont bonnes, les cristaux pourront être obtenus.
Quantification des protéines par spectrophotométrie. La spectrophotométrie permet de détecter et de quantifier les protéines concentrées. C’est une étape de validation cruciale avant l’analyse structurale : si les conditions sont bonnes, les cristaux pourront être obtenus.
 Mise en condition pour cristallisation. La protéine est mélangée à des solutions spécifiques, puis placée sur des plaques hermétiquement fermées. L’objectif : obtenir des cristaux, une forme solide qui fige la protéine pour faciliter l’observation de sa structure atomique par la suite.
Mise en condition pour cristallisation. La protéine est mélangée à des solutions spécifiques, puis placée sur des plaques hermétiquement fermées. L’objectif : obtenir des cristaux, une forme solide qui fige la protéine pour faciliter l’observation de sa structure atomique par la suite.
 Analyse des images de diffraction. La structure de la protéine est révélée par diffraction des rayons X, sur les lignes Proxima du synchrotron SOLEIL5. Le faisceau traverse le cristal et se diffracte, produisant un motif ponctué (écran du milieu). En combinant ces images, les scientifiques calculent la position de chaque atome et reconstruisent la forme 3D de l’enzyme.
Analyse des images de diffraction. La structure de la protéine est révélée par diffraction des rayons X, sur les lignes Proxima du synchrotron SOLEIL5. Le faisceau traverse le cristal et se diffracte, produisant un motif ponctué (écran du milieu). En combinant ces images, les scientifiques calculent la position de chaque atome et reconstruisent la forme 3D de l’enzyme.
 Modélisation finale et interprétation. Les structures 3D obtenues sont affinées, visualisées sur ordinateur et parfois imprimées. Ces modèles révèlent les enzymes actrices du cycle de Calvin. Reste à décrypter leurs actions dans le contexte biologique : comprendre comment elles s’assemblent, interagissent et orchestrent les rouages d’un cycle biochimique primordial à la vie sur Terre.
Modélisation finale et interprétation. Les structures 3D obtenues sont affinées, visualisées sur ordinateur et parfois imprimées. Ces modèles révèlent les enzymes actrices du cycle de Calvin. Reste à décrypter leurs actions dans le contexte biologique : comprendre comment elles s’assemblent, interagissent et orchestrent les rouages d’un cycle biochimique primordial à la vie sur Terre.
- 1. Processus par lequel une plante produit sa propre nourriture (glucose) à partir du dioxyde de carbone présent dans l’air, de la lumière du soleil et de l’eau.
- 2. Unités CNRS/Sorbonne Université.
- 3. Protéine accélérant les réactions chimiques d’un organisme.
- 4. Molécule produite à partir de l’ADN transportant l’information génétique nécessaire à la fabrication d’une protéine.
- 5. Installation située à Saint-Aubin, dans le pôle scientifique de Paris-Saclay, qui produit, à l’aide d’accélérateurs de particules, une lumière si brillante qu’elle permet d’explorer la matière jusqu’à l’échelle atomique.



