A la une

Imaginez. Votre royaume corporel pris d’assaut par un agent pathogène. Vos cellules sentinelles sonnent l’alarme en déclenchant l’inflammation. Si la bataille persiste, les forces adaptatives que sont vos lymphocytes interviennent. Les lymphocytes T sont en quelque sorte des stratèges capables de cibler et d’éliminer les cellules infectées qu’ils identifient grâce aux récepteurs de cellules T. Ils génèrent ensuite des clones – une armée de soldats identiques disposant de ce même récepteur – pour lutter contre l’agent pathogène. Les lymphocytes B forgent, quant à eux, des anticorps spécifiques pour neutraliser l’ennemi. Une fois l’infection vaincue, votre citadelle corporelle est renforcée par la mémoire immunitaire. Celle-ci repose sur des lymphocytes T et B qui restent dormants dans votre organisme, mais prêts à réagir plus rapidement en cas de récidive de cet adversaire.
Ces batailles sont monnaie courante dans notre organisme. Si bien que nous possédons un répertoire immunitaire qui liste les milliards d’anticorps et de récepteurs de cellules T à même de reconnaître de nombreux antigènes1. Ce répertoire change au fil du temps en réponse aux vaccinations et à d'autres stimuli externes. Mais comment évolue-t-il pendant une infection ? Cette question marque le point de départ du projet RESP-REP2. « Nous avons développé de nouvelles méthodes de traitement de données de séquençage de ces répertoires afin d’étudier la réponse immunitaire. L’idée était de comprendre de manière statistique, quels récepteurs de cellules T sont impliqués face à quels antigènes au-delà de ce qui était déjà connu », décrit Aleksandra Walczak, chercheuse au Laboratoire de physique de l'ENS (LPENS)3 et copilote du projet.
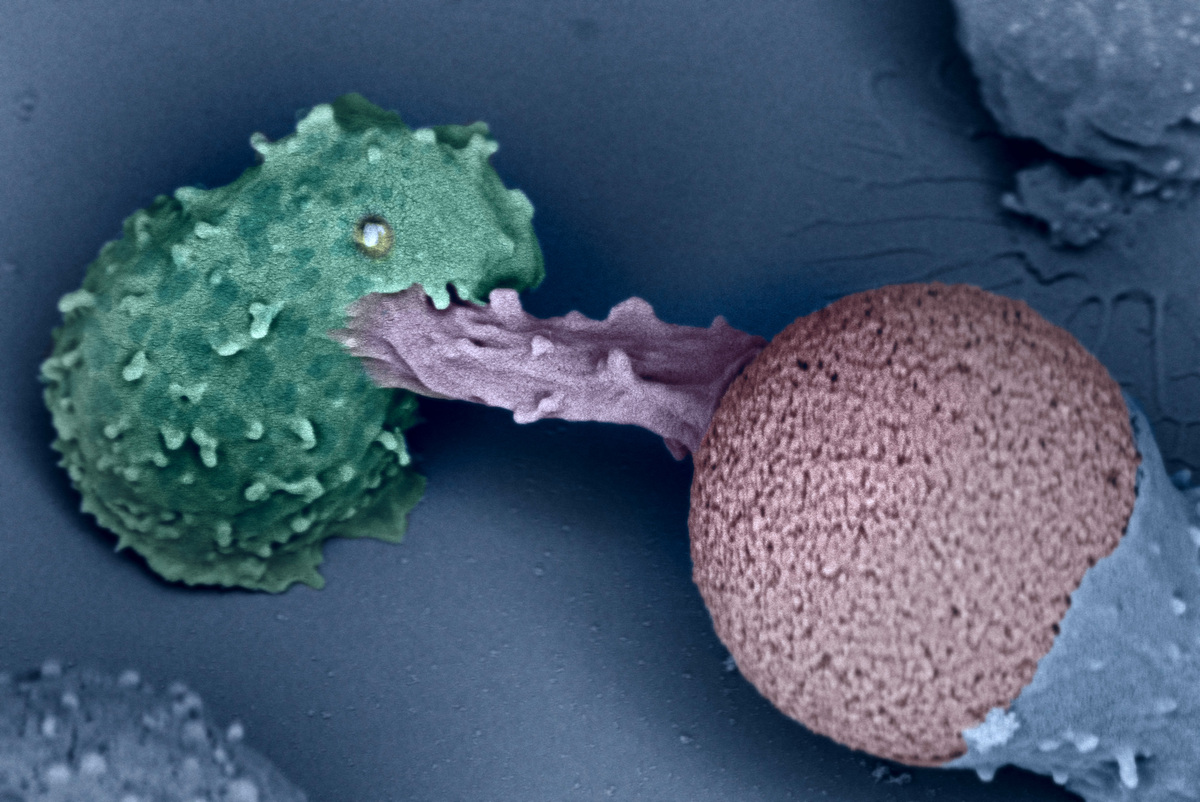 Lymphocyte T (à gauche) en pleine reconnaissance d’un intrus, ici une microbille de plastique (à droite). Cette image a été lauréate du concours « La preuve par l’image » 2019. © Virginie BAZIN / Claire HIVROZ / Julien HUSSON / IPBS / Institut Curie / LadHyX / CNRS Images
Lymphocyte T (à gauche) en pleine reconnaissance d’un intrus, ici une microbille de plastique (à droite). Cette image a été lauréate du concours « La preuve par l’image » 2019. © Virginie BAZIN / Claire HIVROZ / Julien HUSSON / IPBS / Institut Curie / LadHyX / CNRS Images
Un modèle grandeur nature
Avec leurs collègues moscovites, les chercheurs du projet s’intéressent d’abord à un modèle d’infection associé à la vaccination contre la fièvre jaune. Mais peu de temps après, le monde est frappé par la pandémie de COVID-19. Les scientifiques basculent alors leur terrain d’étude sur un vrai modèle grandeur nature. « Nous avons eu accès à des échantillons sanguins de patients vers le pic de leur infection et quelques semaines après. Cela nous a permis de détecter les séquences de récepteurs de lymphocytes T spécifiquement impliqués dans leur réponse immunitaire et leur évolution au cours du temps », explique Thierry Mora, chercheur au LPENS et copilote de RESP-REP. Les chercheurs observent notamment la présence de clones de lymphocytes T spécifiques à ce virus dans la mémoire immunitaire de l’individu trois mois après le pic infectieux.
Cependant, alors que les antigènes et les lymphocytes T diffèrent d’un virus à l’autre, cette étude a démontré une proximité entre la réaction au COVID-19 et d’autres coronavirus. « Certains des clones de lymphocytes T impliqués ici étaient déjà présents dans le compartiment mémoire de certains patients deux ans avant la pandémie. Leur présence pourrait être la manifestation d’une immunité croisée entre différents virus de la même famille. Cette mémoire a peut-être permis à certaines personnes d’avoir une meilleure réponse immunitaire », explique Aleksandra Walczak.
Un répertoire de questions
Notre corps est donc capable de se souvenir d’infections passées, mais pour combien de temps ? « Nous ne savons pas encore et avec le COVID-19, nous avons aussi réalisé que les gens pouvaient avoir une mémoire immunitaire qui ne protège pas forcément contre les infections. Peut-être mitige-t-elle les effets du virus, mais cela n’est pas encore bien compris », rapporte Thierry Mora. Pour la suite, les chercheurs souhaiteraient donc regarder les répertoires d’une plus grande cohorte de patients avec davantage d’échantillons répartis dans le temps. Cela permettrait d’observer les différentes dynamiques de lymphocytes T et leurs liens éventuels avec la gravité de la maladie. Ces informations aideraient plus largement à comprendre pourquoi les réactions peuvent être très différentes d’un malade à l’autre.
Par ailleurs, la méthode développée ici ne saurait se limiter au cadre du COVID-19. Par exemple, pourquoi certains vaccins réalisés pendant l’enfance nous protègent à vie, alors que d’autres doivent être répétés régulièrement ? « Aujourd’hui, il n’y a pas de théorie générale pour expliquer les rythmes de vaccination. Nous pensons qu’une meilleure compréhension des réponses immunitaires et notamment du fonctionnement des répertoires pourrait aider à mieux comprendre les différents processus de mémoire immunitaire », ajoute Aleksandra Walczak.
Vers un diagnostic automatique et systématique ?
Les résultats obtenus ouvrent la voie à une nouvelle méthode de diagnostic à partir de tests sanguins. Pour le moment, la présence dans le sang des récepteurs identifiés par cette étude signifie qu’une personne a été infectée par le COVID-19. L’intérêt d’une telle analyse pourrait être décuplé si elle s’appliquait à d’autres infections. « Si l’on connaissait les séquences de récepteurs des lymphocytes T spécifiques à plein d’agents pathogènes différents, alors il suffirait d’une seule analyse de répertoire sanguin pour déterminer si une personne a une protection contre x ou y virus, obtenir des indications sur son statut vaccinal et plus largement son historique d’infection », décrit Thierry Mora. Pour cela, de nouvelles données doivent être captées par des praticiens en vue d’automatiser les diagnostics.
Ces résultats pourraient également servir au développement d’immunothérapies. En effet, un des enjeux de ces dernières est de stimuler les lymphocytes T spécifiques à des néoantigènes – des parties du génome ayant subi une mutation comme dans le cas d’un cancer. Identifier les cellules T et les récepteurs capables de reconnaître ces cellules anormales pourrait servir au développement de vaccins chargés de réactiver une défense immunitaire contre les tumeurs.
--------------------
Ces recherches ont été financées en tout ou partie, par l’Agence Nationale de la Recherche (ANR) au titre du projet ANR-RESP-REP-AAPG2019. Cette communication est réalisée et financée dans le cadre de l’appel à projets Science Avec et Pour la Société - Culture Scientifique Technique et Industrielle pour les projets JCJC et PPRC des appels à projets génériques 2018-2019 (SAPS-CSTI-JCJ et PRC AAPG 18/19).



