A la une

Essentiel mais encore énigmatique, le complexe SAGA intrigue les biologistes. À Montpellier, Dom Helmlinger et son équipe s’attaquent à une question clé : quels gènes ce cofacteur de la transcription régule-t-il ? Tel était l’objet du projet ANR COACTIVATOR, au cours duquel les chercheurs ont recouru à des approches expérimentales innovantes.
Du haut de ses 19 sous-unités, le complexe SAGA, acronyme de « Spt-Ada-Gcn5 acétyltransférase », interagit avec la machinerie cellulaire chargée de transcrire les gènes en ARN messagers, mais sans se lier directement à l’ADN. Par ses activités de modification des histones, protéines associées à l’ADN, il favorise une décondensation locale de la chromatine. C’est cet état relâché qui permet le recrutement des facteurs de transcription, et donc l’expression des gènes.
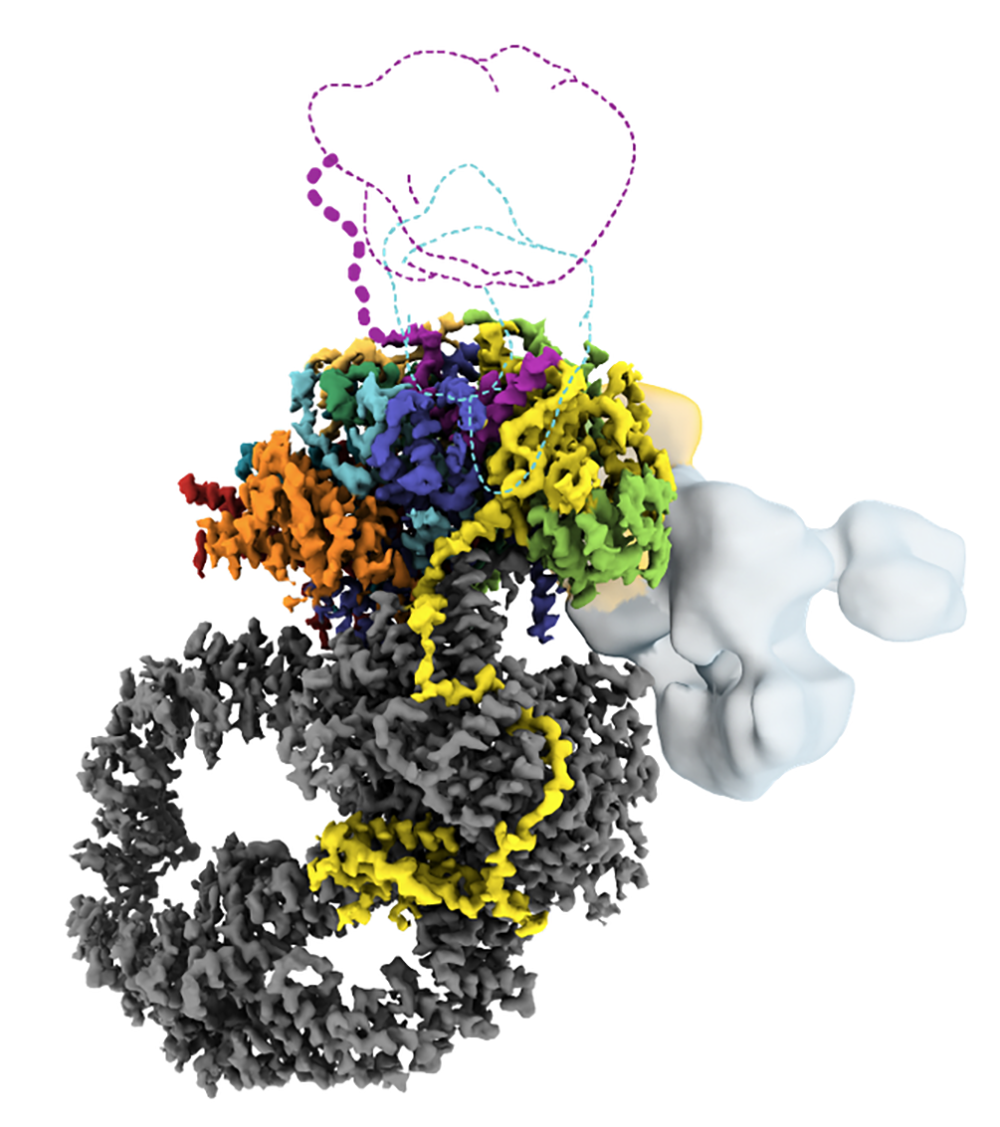 Modèle 3D du complexe SAGA purifié de cellules humaines et visualisé par cryo-microscopie électronique. Certaines sous-unités sont bien visibles et colorées selon leur identité, alors que d’autres ne sont pas suffisamment résolues pour être identifiées, probablement car leur conformation est très mobile. © D. Herbst et al., Nature Structural & Molecular Biology, 2021 DOI: 10.1038/s41594-021-00682-7
Modèle 3D du complexe SAGA purifié de cellules humaines et visualisé par cryo-microscopie électronique. Certaines sous-unités sont bien visibles et colorées selon leur identité, alors que d’autres ne sont pas suffisamment résolues pour être identifiées, probablement car leur conformation est très mobile. © D. Herbst et al., Nature Structural & Molecular Biology, 2021 DOI: 10.1038/s41594-021-00682-7
Or bien que le fonctionnement de SAGA soit de mieux en mieux connu, nul ne sait réellement quels gènes il régule. Un flou qui agace le chercheur montpelliérain Dom Helmlinger, responsable de l’équipe « Régulation de l’expression des gènes » au Centre de recherche en biologie cellulaire de Montpellier1, pourtant grand connaisseur de SAGA. « Si on vient me demander ‘quels sont les gènes qui dépendent de SAGA, à quelle fonction biologique vais-je toucher si j’inactive ce complexe ?’, et bien en fait… je ne le sais pas ! Comme ces facteurs n’ont pas de sites de liaison sur l’ADN identifiés, il m’est impossible de dresser une liste des gènes qu’il cible. C’est comme si je travaillais sur une enzyme, mais sans connaître ses substrats ! ».
Alors que les techniques de laboratoire ne cessent de se sophistiquer, et que le séquençage est désormais qualifié de ‘haut débit’, ce dilemme devient toujours plus criant, et pas uniquement pour SAGA. Plusieurs équipes se sont déjà attelées à ce problème, avec un succès mitigé, même lors de travaux portant sur plusieurs centaines de facteurs et cofacteurs. Plusieurs d’entre eux, menés aussi bien chez la levure, la mouche, la souris que dans des cellules humaines, ont tenté de comparer d’une part les gènes sous dépendance d’un (co)facteur de transcription, d’autre part la localisation de ces facteurs le long de l’ADN. Dans un monde idéal, sans biais expérimentaux, la concordance devrait être facilement visible. Or il n’en est rien : en moyenne, le taux de recoupement entre les deux ensembles de gènes ne dépassait pas quelques pourcents !
Quels gènes activés par SAGA ?
C’est de ce constat qu’est né le projet ANR COACTIVATOR, lancé en 2021 par l’équipe de Dom Helmlinger, en collaboration avec des chercheurs de l’Institut de génétique, biologie moléculaire et cellulaire2. Son objectif était de déterminer l’ensemble des gènes régulés par le complexe SAGA, tout en visualisant sa localisation sur le génome. Originalité de ce projet, les chercheurs ont identifié trois écueils majeurs rencontrés lors de précédentes études, leur apportant autant de réponses techniques.
Primo, l’équipe s’est penchée sur les moyens d’inactiver au mieux le complexe SAGA -une opération qui permet, par comparaison avec un contrôle, d’identifier les gènes qu’il active. Avec les mutants utilisés jusqu’alors, l’effet de la mutation ne se fait sentir qu’au bout « de 48 à 72 heures » au plus tôt, explique Dom Helmlinger. Or il est désormais avéré que ce délai est suffisamment long pour que les cellules s’adaptent, éventuellement en corrigeant partiellement l’effet de la mutation -au risque de voiler ses effets.
Afin d’éviter ce phénomène de compensation, l’équipe montpelliéraine a recouru à un mutant dit ‘conditionnel’, reposant sur la technique du degron. Fixée à une protéine d’intérêt, ce petit motif protéique, sans effet majeur en conditions normales, entraîne la dégradation de la protéine lorsque la cellule est exposée à un signal -le plus fréquemment utilisé est l’ajout d’auxine, une hormone végétale. Avantage majeur de cette technique, la dégradation est rapide (quelques heures), réversible et spécifique de la protéine ciblée.
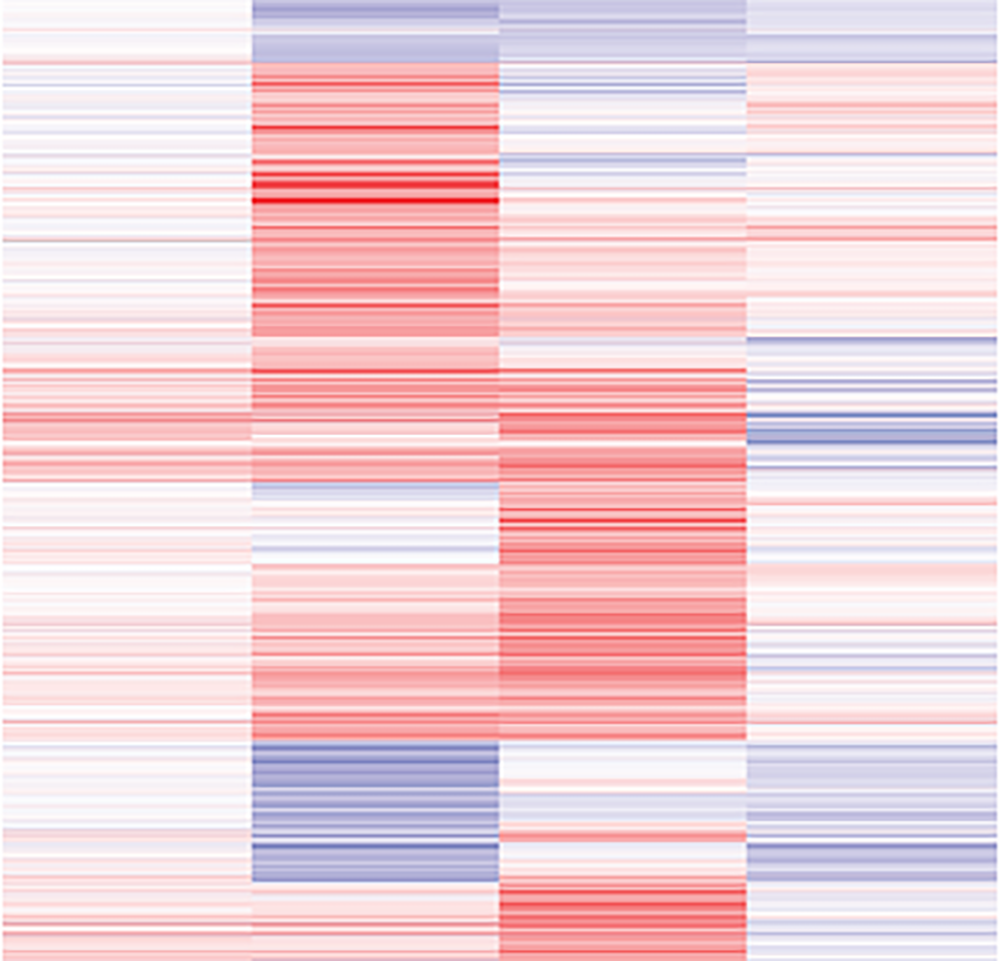 Représentation graphique de l’effet d’une perte de SAGA sur le niveau de transcription de plusieurs centaines de gènes. Chaque colonne montre une condition expérimentale différente et chaque ligne montre le niveau d’un ARN, coloré selon que celui-ci augmente (rouge), baisse (bleu) ou ne change pas (blanc). © Dom Helmlinger
Représentation graphique de l’effet d’une perte de SAGA sur le niveau de transcription de plusieurs centaines de gènes. Chaque colonne montre une condition expérimentale différente et chaque ligne montre le niveau d’un ARN, coloré selon que celui-ci augmente (rouge), baisse (bleu) ou ne change pas (blanc). © Dom Helmlinger
Les ARNm identifiés à la source
Deuxio, les chercheurs ont évalué l’effet transcriptionnel des mutants SAGA non pas en analysant l’ensemble des ARNm présents dans la cellule à l’instant t, mais seulement ceux en cours de transcription. La nuance est de taille : « il ne s’agit pas de regarder l’ARN à l’équilibre, mais le taux de synthèse. Car du fait que la cellule s’adapte à un déséquilibre , elle peut réajuster son niveau de dégradation des ARN. Il se peut donc que le niveau d’ARN à l’équilibre ne change pas », explique Dom Helmlinger.
Pour éviter cela, l’équipe a recouru à un marquage métabolique combiné à la ‘click chemistry’. Cette dernière a été distinguée par le prix Nobel de chimie en 2022. Appliquée à la biologie moléculaire, cette ‘chimie click’ repose sur l’emploi de nucléotides modifiés, incorporés dans l’ARN en cours de transcription. Grâce à ce marquage chimique, il est ensuite possible d’isoler les ARN récemment synthétisés de l’ensemble de ceux présents dans la cellule. Ces ARN, en très faible quantité, peuvent ensuite être identifiés et quantifiés par séquençage haut-débit.
Troisième innovation utilisée par l’équipe, une première pour une équipe montpelliéraine, le ‘CUT&RUN’, qui vise à localiser un (co)facteur de transcription sur l’ADN. En raison de la complexité de la chromatine, la technique standard engendre un important bruit de fond -d’autant plus élevé lorsqu’il s’agit de complexes, comme SAGA, ne se liant pas directement à l’ADN. Au lieu d’opérer sur l’ensemble de la chromatine, le ‘CUT&RUN’ permet d’isoler spécifiquement les petits fragments d’ADN marqués par l’empreinte du complexe SAGA. Jusqu’alors, « il s’agissait d’extraire péniblement une aiguille d’une botte de foin. Avec le ‘CUT&RUN’, l’aiguille sort d’elle-même, et on peut jeter la botte de foin », explique Dom Helmlinger.
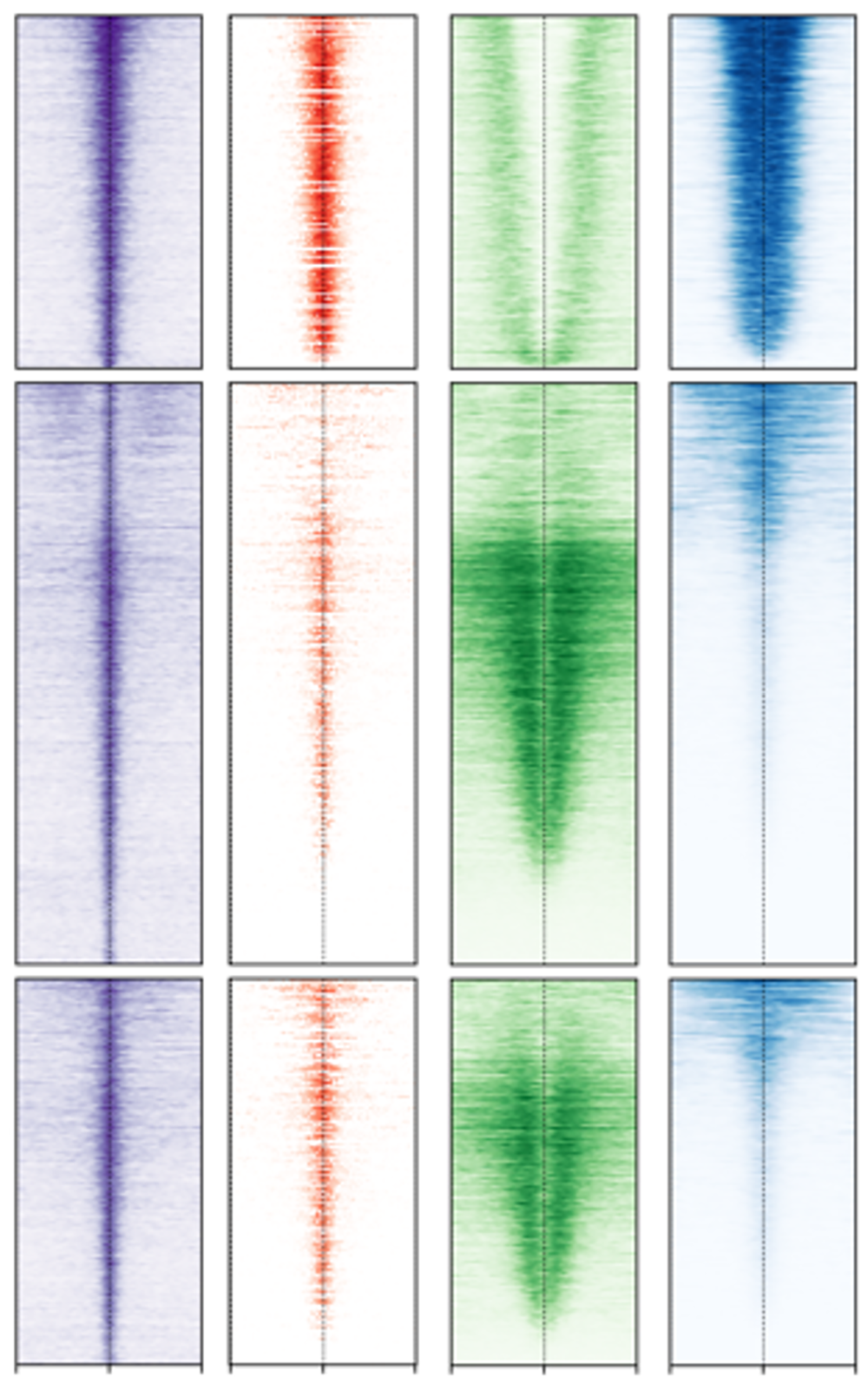 Illustration des profils de localisation de SAGA à la chromatine (en violet), de l’accessibilité de l’ADN (orange), d’une marque d’histone enrichie aux ‘enhancers’ (vert) et d’une marque d’histone enrichie aux promoteurs (bleu) identifiées par séquençage haut-débit. Chaque ligne correspond à une région du génome humain régulant la transcription, dont plus de 15 000 sont montrées sur ces cartes © Dom Helmlinger
Illustration des profils de localisation de SAGA à la chromatine (en violet), de l’accessibilité de l’ADN (orange), d’une marque d’histone enrichie aux ‘enhancers’ (vert) et d’une marque d’histone enrichie aux promoteurs (bleu) identifiées par séquençage haut-débit. Chaque ligne correspond à une région du génome humain régulant la transcription, dont plus de 15 000 sont montrées sur ces cartes © Dom Helmlinger
Au-delà des aléas de la recherche, de nouvelles découvertes
Par ces trois approches, l’équipe était solidement outillée pour définir les gènes dépendant de SAGA, ainsi que la localisation de celui-ci sur l’ADN. Mais si les premiers résultats, obtenus sur une lignée de cellules de cancer colorectal, se sont avérés très encourageants, l’affaire s’est corsée par la suite. Dans le cadre de son projet ANR, l’équipe avait opté pour une lignée issue d’un lymphome humain, l’une de celles identifiées comme prioritaires par le consortium international ENCODE3. Or, si les trois techniques employées par l’équipe avaient fait leurs preuves avec la première lignée, elles se sont avérées bien moins efficaces avec la nouvelle.
Malgré ces déconvenues, l’équipe a mis en évidence un fait jusqu’alors méconnu : SAGA ne se fixe pas seulement au niveau des séquences promotrices, celles situées immédiatement en amont du gène exprimé. Il est aussi présent au niveau des ‘enhancers’. Situées à plus grande distance des gènes, ces séquences jouent un rôle primordial dans leur expression, grâce à un repliement de l’ADN qui les amène au contact des gènes. L’équipe cherche actuellement à comprendre le rôle joué par SAGA au niveau des ‘enhancers’ : favorise-t-il leur proximité avec le gène, ou bien permet-il un remodelage de la chromatine, et donc l’accès d’autres facteurs de transcription au gène ? A moins qu’il n’agisse de manière plus directe sur la transcription de ces séquences…
Tout en ironisant sur sa « crise de la quarantaine » scientifique, tourmentée mais créative, Dom Helmlinger scrute désormais d’autres horizons. Passionné par la levure Schizosaccharomyces pombe, qu’il a découverte lors de son postdoctorat à Boston, le chercheur s’interroge sur son histoire naturelle, et plus particulièrement sur sa présence, jusqu’alors méconnue… dans le miel ! Comment ce micro-organisme y tolère-t-il une concentration si extrême de sucres, toxique à bien des cellules ? Et quelles voies évolutives, quels mécanismes moléculaires, ont permis à certaines souches de S. pombe, mais pas à d’autres, d’y survivre ? Enfin, la présence de cette levure dans les ressources nutritives des abeilles a-t-elle un impact sur les populations de ce pollinisateur essentiel à l’homme ? Coordinateur d’un nouveau projet ANR, dénommé POMBEE et mené en collaboration avec le Centre d’écologie fonctionnelle et évolutive (CEFE, Montpellier), le chercheur compte bien défricher les grands espaces, encore peu explorés, entre génétique moléculaire, écologie et biologie de l’évolution.
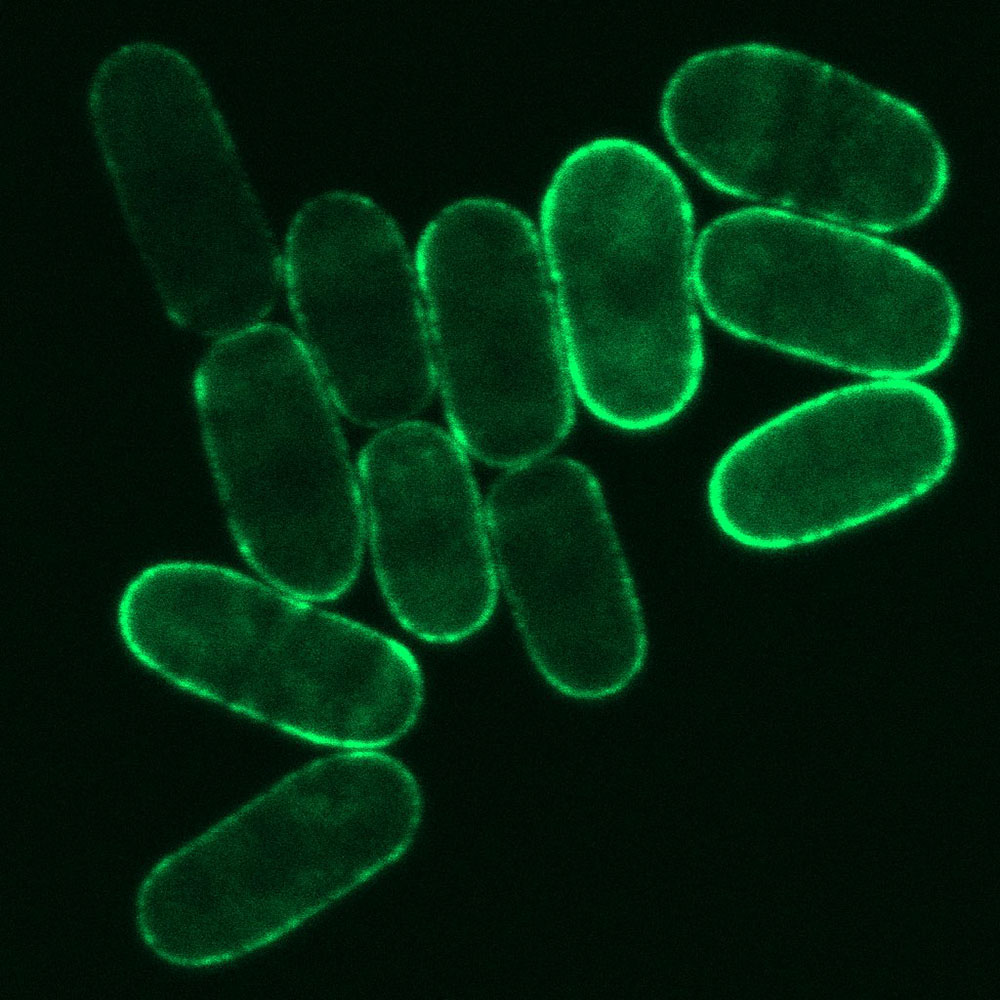 Visualisation de la levure Schizosaccharomyces pombe par épifluorescence d’une souche marquée avec un rapporteur GFP localisé à la périphérie. © Dom Helmlinger
Visualisation de la levure Schizosaccharomyces pombe par épifluorescence d’une souche marquée avec un rapporteur GFP localisé à la périphérie. © Dom Helmlinger
-------------------------------------------------------------------------------------------------------------------------------
Ces recherches ont été financées en tout ou partie, par l’Agence Nationale de la Recherche (ANR) au titre de l'ANR COACTIVATOR - AAPG2020. Cette communication est réalisée et financée dans le cadre de l’appel à projet Science Avec et Pour la Société - Culture Scientifique Technique et Industrielle pour les projets JCJC et PRC des appels à projets génériques 2020 (SAPS-CSTI JCJC et PRC AAPG 20).
- 1. CRBM – Unité CNRS / Université de Montpellier.
- 2. IGBMC – Unité CNRS / INSERM / Université de Strasbourg.
- 3. Lancé en 2003 dans la foulée du Human Genome Project, le projet ENCODE vise à identifier tous les éléments fonctionnels du génome humain, en particulier ceux ne codant pas pour un gène. Y compris d’un point de vue épigénétique, un niveau de régulation de l’expression des gènes reposant sur des modifications chimiques des protéines histones (acétylation, phosphorylation, etc.) et de l’ADN (méthylation des îlots CpG).



