Vous êtes ici
Un pas de géant pour l'étude des biofilms

Pouvez-vous nous rappeler ce qu’est un biofilm bactérien ?
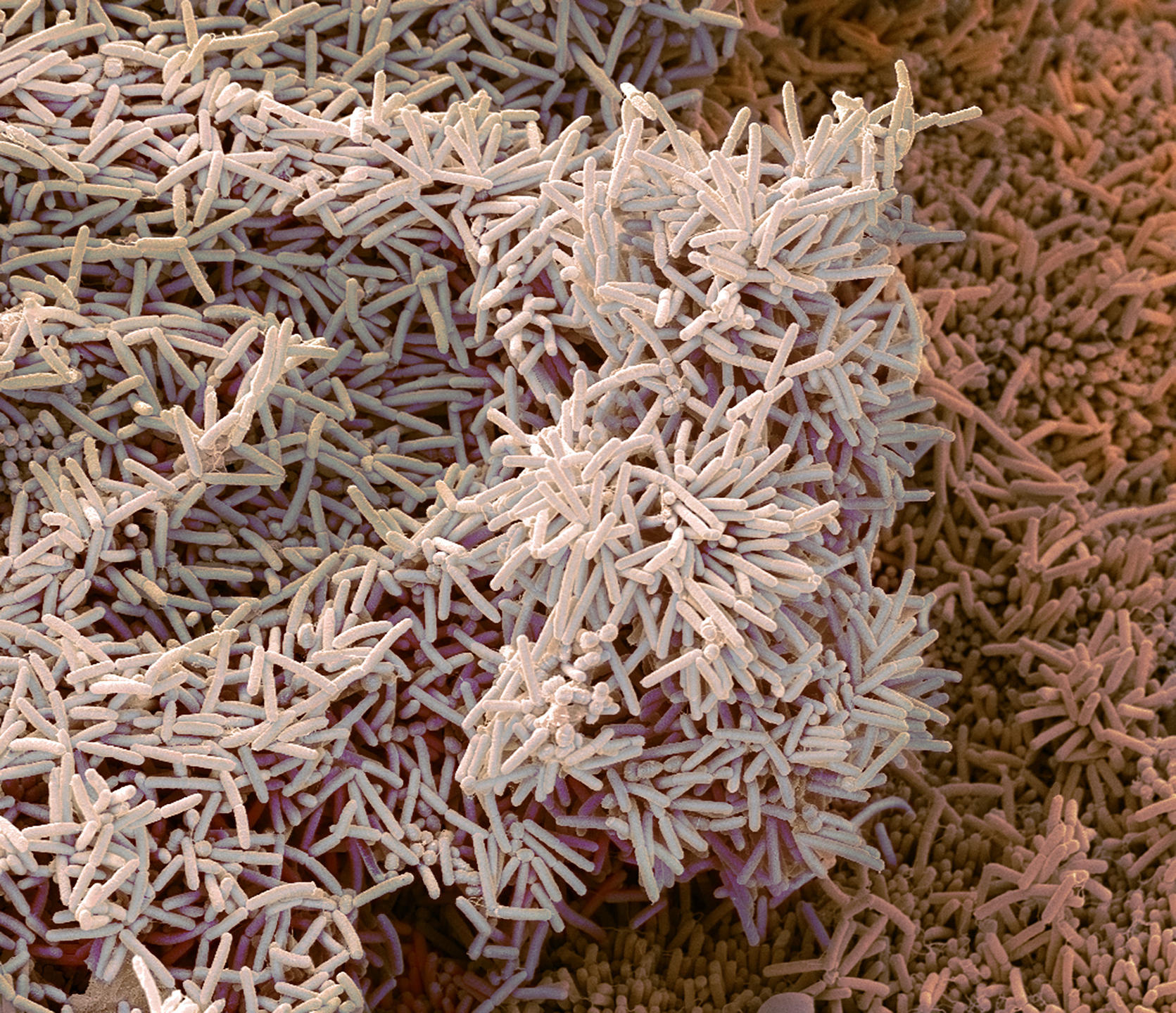
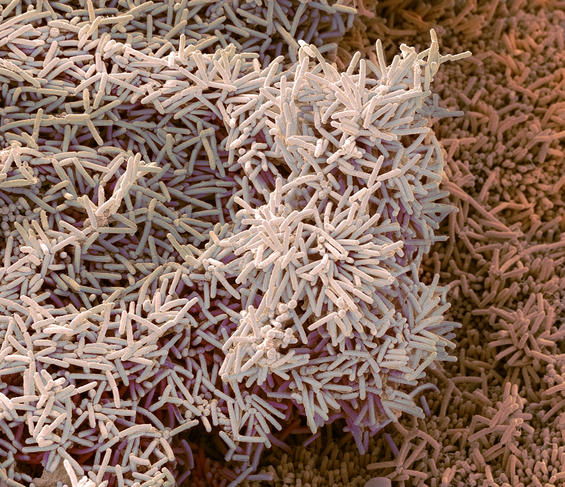
Tâm Mignot1. Il s’agit d’un assemblage de plusieurs centaines à plusieurs millions de bactéries, appartenant à une ou plusieurs espèces, qui adhèrent à une surface et entre elles via la sécrétion de polymères naturels dits « exopolysaccharides ». Ce type de structures existe partout : dans la nature (dans les sols, les océans, sur les végétaux, sur les animaux…), dans l’industrie (au niveau par exemple des filtres des raffineries de pétrole ou des coques de navires) ou encore à l’hôpital (sur du matériel d’injection comme des cathéters, ou sur des prothèses ou des plaies).
Quels intérêts à les étudier ?
T. M. En savoir plus sur les biofilms bactériens est crucial non seulement pour la recherche fondamentale, mais aussi pour la recherche appliquée, notamment médicale, agronomique et environnementale. Concernant la première, un de ses objectifs est d’affiner nos connaissances sur les processus cellulaires et moléculaires via lesquels les biofilms se forment et se dissolvent. Car jusqu’il y a encore quelques décennies, elle s’est surtout focalisée sur l’étude de bactéries individuelles et en culture. Or l’examen des biofilms dans la nature a révélé que jusqu’à 80 % des micro-organismes de notre planète vivraient sous cette forme et non isolés…
Quid de l’importance de leur étude en recherche appliquée ?
T. M. Au niveau médical, l’analyse des molécules impliquées dans la dissolution des biofilms bactériens pourrait mener à de nouveaux traitements antimicrobiens plus efficaces contre les pathogènes se développant à l’hôpital. Côté environnement, la recherche sur ces structures pourrait aider à favoriser la formation de biofilms bénéfiques pour la nature, certains étant capables de fixer ou de dégrader certains polluants (métaux lourds, hydrocarbures…) ou le carbone du dioxyde de carbone de l’air. Enfin, dans le domaine agronomique, une compréhension fine des processus de formation des biofilms pourrait permettre de stimuler leur développement au niveau des racines des végétaux : en effet, certains peuvent optimiser la croissance des plantes en favorisant la fixation de l’azote du sol dans leurs racines.
Qu’est-ce qu’au juste MiSiC ?
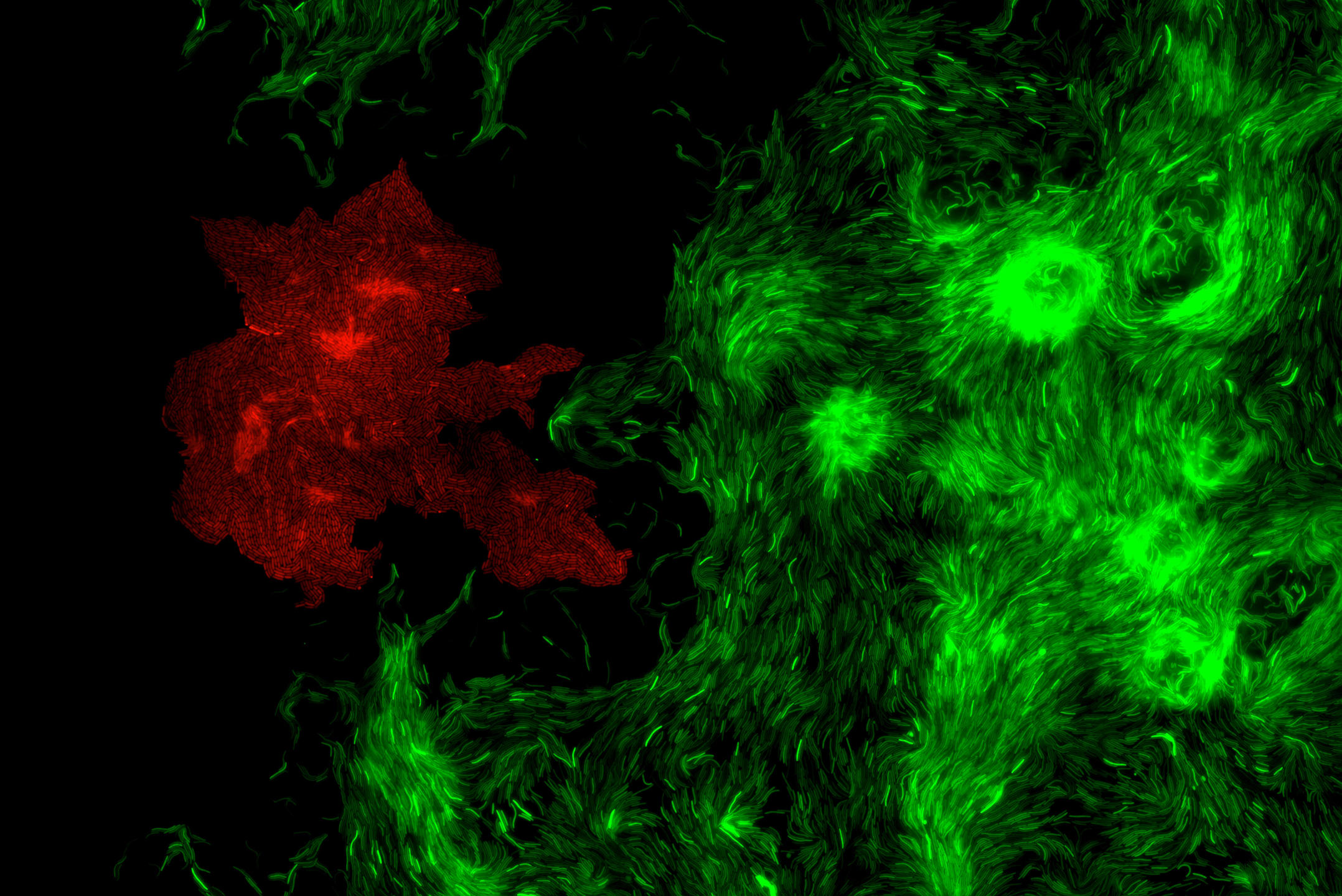
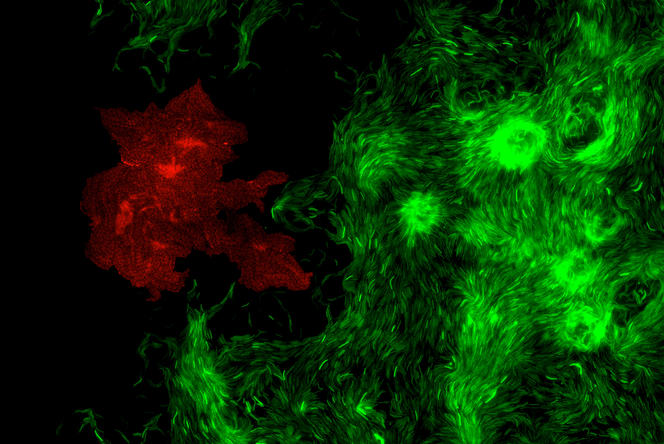
T. M. C’est un logiciel d’analyse d’images microscopiques, qui permet l’étude de la structure d’une communauté microbienne dense, autrement dit d’un biofilm bactérien ; MiSiC étant l’abréviation de l’anglais « Microbial Segmentation in dense Colonies », qui signifie « segmentation microbienne en colonies denses ». Plus précisément, notre système peut distinguer les différentes espèces d’un biofilm bactérien et dénombrer le nombre de bactéries de chacune de ces espèces. Téléchargeable gratuitement et installable sous l’interface d’analyse d’images Napari, il est destiné à tous les chercheurs qui étudient les bactéries en colonie ou en biofilms – pourvu que ces derniers soient en deux dimensions, autrement dit plans ; et non en 3D, avec des cellules empilées.
Qu’apporte de plus cet outil par rapport à ceux déjà disponibles ?
T. M. Comme nous l’avons montré lors de travaux publiés en septembre 20212, MiSiC est compatible avec non pas un seul mais tous les types d’images microscopiques : en « contraste de phase » – où les cellules apparaissent en noir sur un fond clair –, en « fluorescence » – où les cellules sont fluorescentes sur fond noir –, et en mode « bright field » – où les cellules brillent sur fond noir. Ensuite, il est applicable à un grand nombre d’espèces bactériennes ; et en cas de biofilms formés de plusieurs espèces, il permet de distinguer celles-ci via des couleurs différentes. Mais surtout, MiSiC permet de dénombrer de façon fiable des millions de bactéries en communauté, alors qu’il était difficile d’en dénombrer ne serait-ce qu’un millier dans ces conditions. De quoi accélérer la recherche sur les biofilms bactériens.
Comment fonctionne-t-il ?
T. M. En cherchant sur l’image microscopique analysée tous les objets ayant une forme de bâtonnet arrondi aux extrémités. En effet, lors du développement de ce système, Swapnesh Panigrahi, expert en intelligence artificielle dans notre équipe, et Leon Espinosa, spécialiste en analyse pour la microbiologie, ont eu le coup de génie de comprendre que les différents types d’images microscopiques peuvent tous être analysés de façon équivalente, si on demande au système de reconnaître non pas des différences d’intensités – comme le font les autres outils – mais des formes. Or beaucoup de bactéries ressemblent à des bâtonnets arrondis aux extrémités. D’où l’idée d’entraîner notre logiciel à apprendre à identifier ce profil en le lui montrant plusieurs milliers fois. C’est l’approche du deep learning, ou apprentissage profond, un type d'intelligence artificielle qui s'appuie sur un réseau de neurones artificiels s'inspirant du cerveau humain.

Comment est née l’idée d’inventer votre logiciel ?
T. M. Elle est née au cours de nos recherches sur une bactérie du sol très intéressante : Myxococcus xanthus, qui a pour particularité d’être un micro-organisme prédateur, capable de coopérer avec ses congénères pour détecter, envahir et tuer collectivement des colonies d’autres bactéries proies, pour s’en nourrir. De fait, M. xanthus constitue un modèle d’étude très puissant pour explorer le processus de prédation chez les bactéries. Sachant que celui-ci est soupçonné d’être, comme chez les mammifères, un élément majeur pour l’équilibre des écosystèmes – car indispensable pour réguler les populations de proies. Or pour analyser finement les mécanismes moléculaires sous-tendant le comportement de prédation de M. xanthus, il était indispensable de disposer d’un outil permettant de voir ce qui survient au niveau de chacune des cellules d’un biofilm formé de millions de bactéries d’espèces différentes. Comme ce système n’existait pas, il a fallu l’inventer !
Déjà de premières avancées grâce à cet outil ?
T. M. Oui. Lors d’une étude publiée deux jours après celle concernant MiSiC3, nous avons découvert qu’au niveau moléculaire, les bactéries M. xanthus tuent leurs proies à l’aide d’une machinerie moléculaire complexe : au contact des proies, elles assemblent un appareil constitué d’une dizaine de protéines, lequel leur permet d’injecter une toxine qui fait exploser les proies. Reste maintenant à identifier cette toxine pour finir d’assembler toutes les pièces du puzzle ! ♦
- 1. Directeur de recherche CNRS au Laboratoire de chimie bactérienne (unité CNRS/Aix-Marseille Université).
- 2. Swapnesh Panigrahi et al. Elife. 9 septembre 2021. doi: 10.7554/eLife.65151.
- 3. Sofiene Seef et al. Elife. 10 septembre 2021. doi: 10.7554/eLife.72409.
Voir aussi
Auteur
Journaliste scientifique freelance depuis dix ans, Kheira Bettayeb est spécialiste des domaines suivants : médecine, biologie, neurosciences, zoologie, astronomie, physique et nouvelles technologies. Elle travaille notamment pour la presse magazine nationale.