A la une

Les symptômes sont connus et décrits depuis l’Antiquité, pourtant, la tuberculose sévit encore avec plusieurs millions de morts par an (1,5 M en 2020). Le bacille de Koch – autre nom donné à la bactérie d’après son découvreur, Robert Koch – établit une infection pulmonaire et durable dans les lésions complexes pulmonaires. Au sein de l’Institut de chimie et de biologie des membranes et des nano-objets de Bordeaux1 (CBMN), une équipe de scientifiques s’intéresse à combiner différentes techniques d’imagerie pour mieux comprendre la maladie et améliorer les effets des antibiotiques. Un des objectifs scientifiques est de quantifier et de localiser plusieurs antibiotiques efficaces contre la tuberculose, comme la Clofazimine (CFZ), la Rifampicine (RIF), ou l’Éthambutol (ETM).
Photos © Gautier Dufau / CBMN / CNRS

Les tissus biologiques infectés par Mycobacterium Tuberculosis, bactérie responsable de la tuberculose, puis désactivé par rayonnement gamma sont reçus dans le laboratoire. Ils représentent comme une photographie d’un stade de la maladie à un instant t. Sectionnés en de très fines coupes par un cryomicrotome (coupe à très basse température), observable ici, elles permettront des analyses intégrant plusieurs familles de biomolécules (comme l’analyse des lipides, ainsi que des antibiotiques).

Une fois les échantillons sectionnés en très fines coupes, celles-ci sont conservés et préservés dans un congélateur à -80° jusqu’à analyse.

Le microscope assure la suite de l’analyse de la coupe préalablement faite grâce au cryomicrotome. L’idée étant de visualiser très nettement les structures d’intérêt à être analyser par imagerie par spectrométrie de masse MALDI. Des cellules immunitaires sont observées, telles que des macrophages, localisées au niveau des tissus de poumons traités.
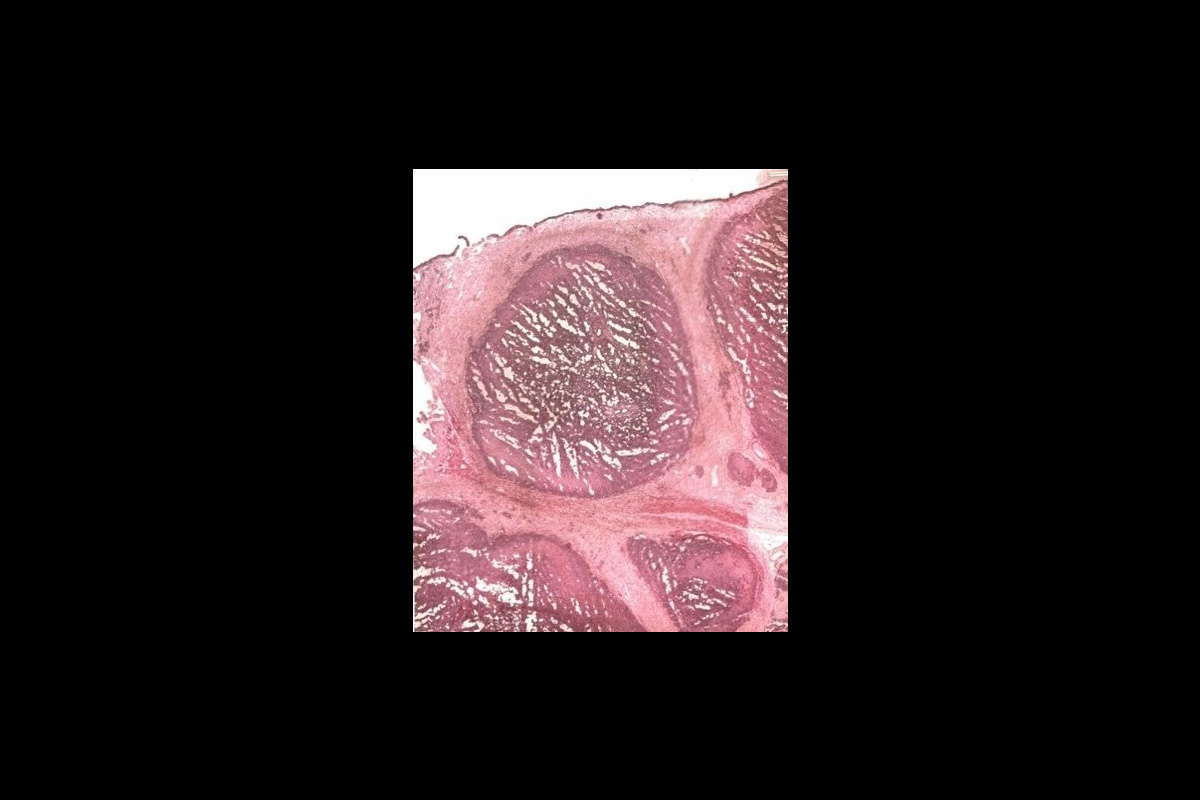
Afin d’avoir une appréhension fine de l’environnement de ces lésions tuberculeuses, des tissus ayant les mêmes propriétés que l’organisme malade sont obtenus grâce des colorations histologiques. Celles-ci permettent de pouvoir identifier les structures anatomiques d’un organe.


Dépôt d’une petite couche de molécules sur l’échantillon des tissus préparés au préalable. Celle-ci va permettre d’optimiser la technique MALDI (Matrix-Assisted Laser Desorption/Ionization). La matrice MALDI agit comme substance chimique qui absorbe l’énergie du laser et la transmet aux molécules du tissu.

Avec cette machine singulière, les scientifiques réalisent de l’imagerie par spectrométrie de masse MALDI. Cette technique physique d’analyse rend possible l’identification ainsi que la localisation de molécules grâce à la mesure de leur masse exacte. En d’autres termes il s’agit ici à la fois d’une balance et d’un GPS moléculaire. La particularité de celle-ci est qu’elle opère à pression atmosphérique, ce qui rend l’analyse plus en adéquation avec l’analyse des tissus biologiques. En optimisant ainsi les paramètres d’acquisition, une série de biomolécules liées la tuberculose sont localisées et quantifiées : antibiotiques, lipides, etc., et délivrent de précieuses informations quant à son traitement.
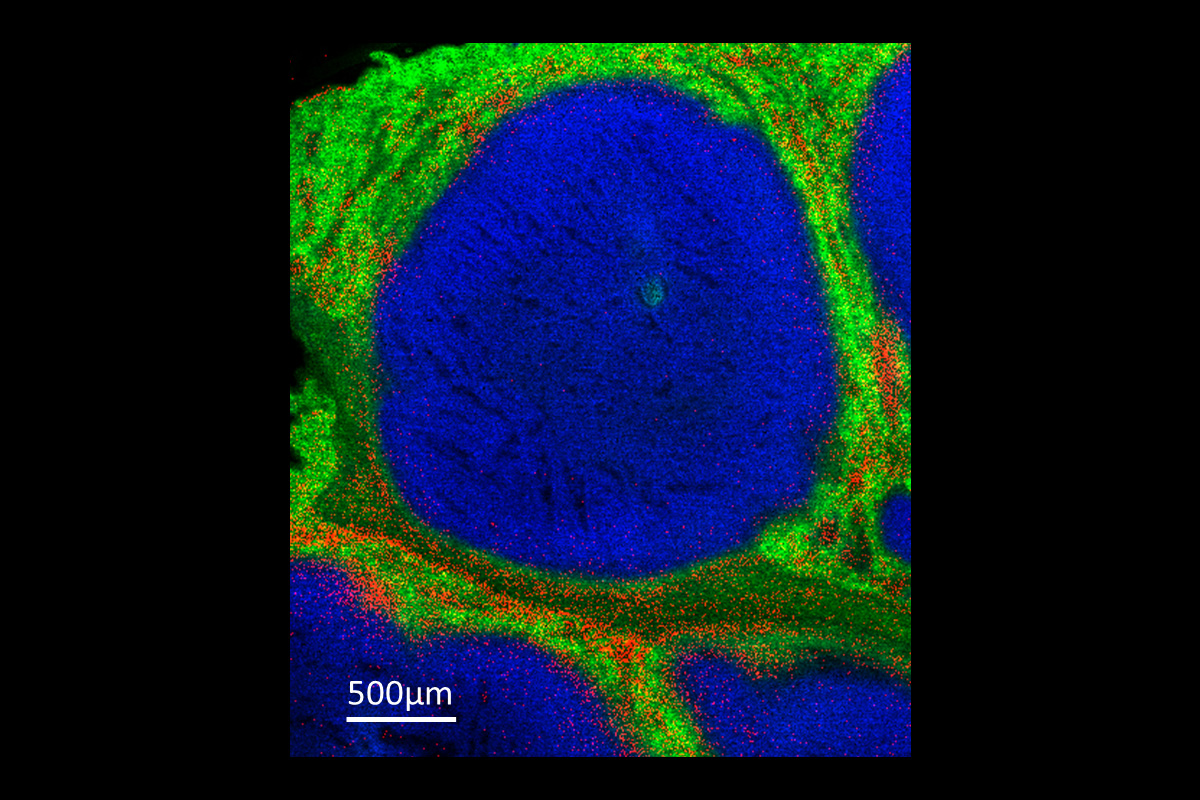
Ici est observée une partie du poumon d’un animal infecté par Mycobacterium Tuberculosis, puis traité avec les antibiotiques orientés contre la tuberculose. Cette image MALDI a été acquise avec une taille de pixel de 5 µm. Nous y retrouvons l’antibiotique CFZ en rouge, dont l’imagerie par spectrométrie de masse a permis de révéler la distribution spatiale et la quantité « exacte » dans les macrophages et les macrophages spumeux. Ces observations ont été confirmé par l’utilisation d’une préparation histologique, nous permettant de corréler les images des antibiotiques ou autres molécules avec les structures anatomiques observées. La finalité de ce projet est de mieux comprendre les effets des combinaisons d’antibiotiques et des durées de traitement sur la tuberculose.
L’ensemble des techniques et technologies présentes au sein de ce projet de recherche s’insèrent dans une lignée prometteuse dont l’ambition est d’enrichir significativement notre connaissance de la tuberculose et de ses traitements. L’équipe s’intéresse davantage, dans un second temps, à la partie bio-informatique qui comprend l’élaboration d’un logiciel permettant d’automatiser le traitement des images obtenues par différentes techniques d’observation physiques et biologiques, vrai challenge pour améliorer et préciser l’interprétation des résultats.
----------------------------------------------------------------------------
Ces recherches ont été financées en tout ou partie par l’Agence Nationale de la Recherche (ANR) au titre du projet « Imagerie multimodale combinant la spectroscopie Raman et la spectrométrie de masse appliquée à la tuberculose – MultiRaMaS». Cette communication est réalisée et financée dans le cadre de l’appel à projets Sciences Avec et Pour la Société – Culture Scientifique Technique et Industrielle.
- 1. Unité CNRS, Bordeaux INP, Université de Bordeaux



