A la une

La génomique est la branche de la biologie qui étudie l’ensemble du matériel génétique d’un organisme, codé dans son ADN. L’analyse et le décodage de ce génome permettent aujourd’hui des avancées en médecine, en biologie, en écologie ou dans les biotechnologies. Des algorithmes d’intelligence artificielle, objet d’intenses recherches, facilitent la recherche spécifique de séquences particulières dans cet immense « livre génétique » et l’interprétation de leur rôle dans l’histoire du vivant.
 © Growtika sur Unsplash
© Growtika sur Unsplash
L’ADN est constitué de « lettres » chimiques appelées nucléotides, symbolisées par les lettres A, T, G et C. Associées par paires, leur succession forme une unique et très longue molécule dont certaines parties sont dites codantes (transcrites en ARN messager puis traduites en protéines) ou non codantes (non transcrite en ARN ou non traduites ensuite). L’un des objectifs de la recherche contemporaine est de déterminer quelles parties de cette immense séquence de lettres sont responsables, par exemple, de la résistance de certaines bactéries ou de l’apparition de maladies génétiques chez l’humain, comme la mucoviscidose ou l’hémophilie.
« Mais l’étude du génome, nous dit Laurent Jacob, chercheur en intelligence artificielle pour la génomique au Laboratoire de biologie computationnelle, quantitative et synthétique1 en collaboration avec des membres du Laboratoire de biométrie et biologie évolutive2, permet aussi de comprendre l’évolution des espèces vivantes. Le principe, ici, est de chercher une séquence, un gène, présent chez toutes les espèces étudiées (légèrement différent d’une espèce à l’autre, Nda) et d’essayer, à partir de l’observation de ces gènes, de reconstruire l’histoire du vivant qui a abouti à ces espèces. Cette histoire est visualisée sous la forme d’un arbre qui montre comment les espèces sont apparues et ont évolué. C’est l’arbre phylogénétique. »
Reconstituer l’arbre du vivant grâce à l’IA
Cette branche de la biologie est appelée « biologie évolutive ». Elle s’intéresse à la reconstruction de ces arbres phylogénétiques, qui retracent l’évolution de différentes espèces au cours du temps à partir d’ancêtres communs d’où elles sont issues.
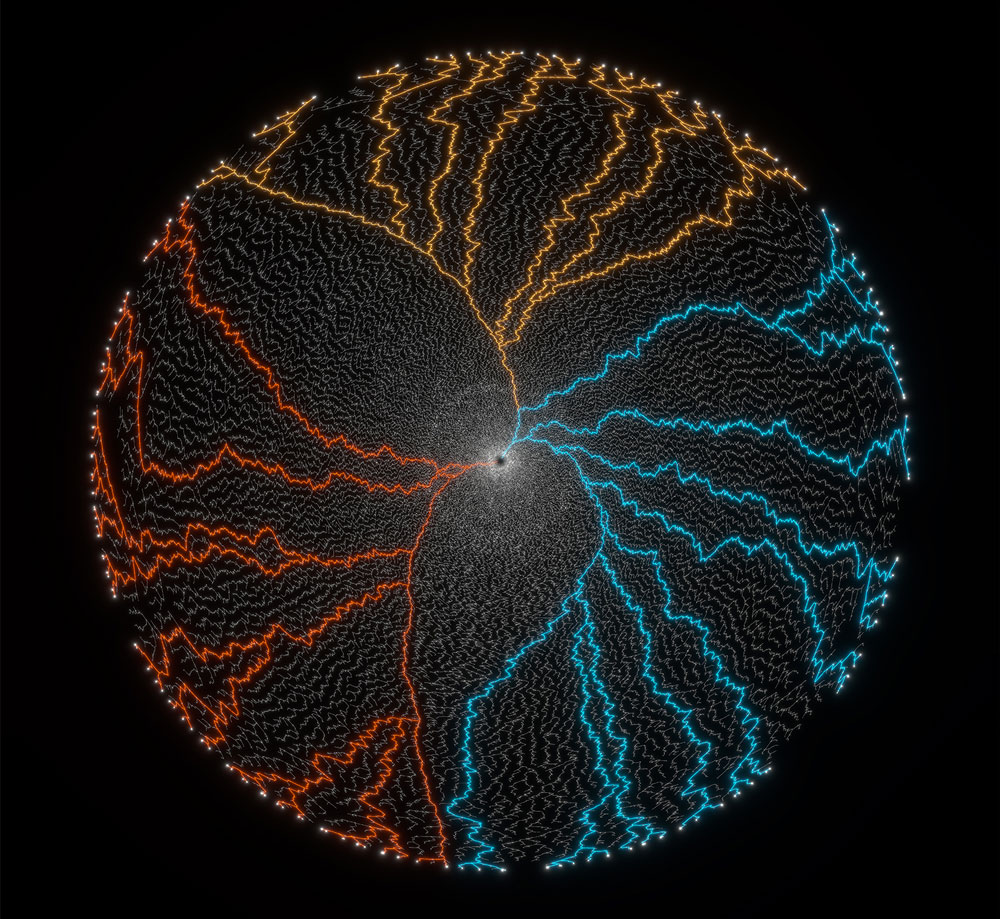 Représentation de l’arbre du vivant mettant en valeur l’importance numérique des espèces éteintes. Les lignées menant aux espèces actuelles (présentées en périphérie du disque) sont colorées, chaque couleur représentant un domaine du vivant (bactéries, archées, eucaryotes). Les lignées aujourd’hui éteintes sont représentées en gris. Le centre du cercle représente l’ancêtre commun de toutes les espèces actuelles, LUCA (pour Last Universal Common Ancestor). (© Alex Andrix / Damien De Vienne / LBBE / CNRS Images)
Représentation de l’arbre du vivant mettant en valeur l’importance numérique des espèces éteintes. Les lignées menant aux espèces actuelles (présentées en périphérie du disque) sont colorées, chaque couleur représentant un domaine du vivant (bactéries, archées, eucaryotes). Les lignées aujourd’hui éteintes sont représentées en gris. Le centre du cercle représente l’ancêtre commun de toutes les espèces actuelles, LUCA (pour Last Universal Common Ancestor). (© Alex Andrix / Damien De Vienne / LBBE / CNRS Images)
L’idée centrale est que ces espèces ont évolué à partir d’un ancêtre commun, suivant des événements de spéciation, produisant plusieurs sous-espèces possédant des versions différentes de ces gènes.
Pour reconstruire cette histoire du vivant à partir des gènes présents dans les espèces contemporaines, l’analyse des génomes peut se faire par « inférence ». Cette technique se fonde sur un modèle probabiliste qui décrit comment une séquence choisie dans le génome des différentes espèces étudiées a eu le plus de chances d’évoluer au cours du temps, en cherchant l’arbre phylogénétique qui rend les séquences observées les plus probables (on dit qu’il maximise la vraisemblance).
La spécificité de l’approche présentée ici est d’entraîner un réseau de neurones à prédire cet arbre phylogénétique à partir des séquences génétiques observées. Elle permet de faire de l’inférence sous des modèles probabilistes plus réalistes, pour lesquels on ne saurait pas maximiser la vraisemblance par le calcul.
Appliquée sur des espèces animales ou végétales, cette méthode permet d’étudier leur apparition, leur évolution, et de lier cette histoire génétique aux grands événements (climatiques, par exemple) qui ont jalonné l’histoire de la vie sur la Terre. Elle permet, par exemple, de comprendre pourquoi certaines périodes de l’histoire ont vu foisonner beaucoup de nouvelles espèces, alors que d’autres ont été très calmes en termes d’évolution.
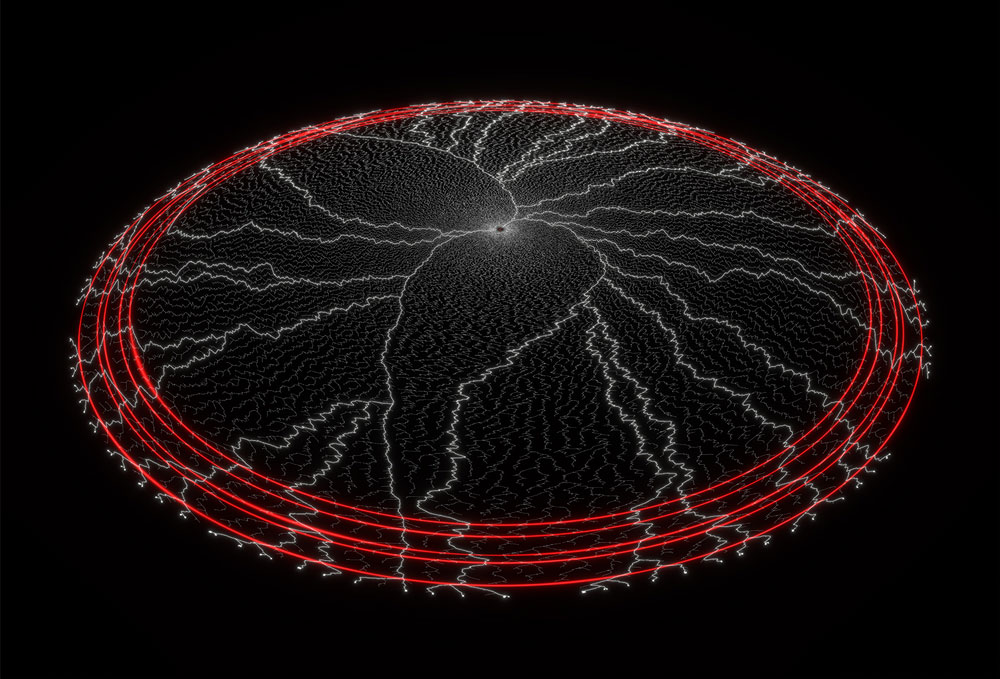 Une représentation de l’arbre du vivant et des grandes extinctions de masse connues. Les lignées menant aux espèces actuelles sont représentées en blanc quand les lignées aujourd’hui éteintes sont grises. Le centre du cercle représente l’ancêtre commun à toutes les espèces actuelles (LUCA). Le temps présent est en périphérie. Les cercles rouges représentent les 5 grandes extinctions massives de biodiversité connues. (© Alex Andrix / Damien De Vienne / LBBE / CNRS Images)
Une représentation de l’arbre du vivant et des grandes extinctions de masse connues. Les lignées menant aux espèces actuelles sont représentées en blanc quand les lignées aujourd’hui éteintes sont grises. Le centre du cercle représente l’ancêtre commun à toutes les espèces actuelles (LUCA). Le temps présent est en périphérie. Les cercles rouges représentent les 5 grandes extinctions massives de biodiversité connues. (© Alex Andrix / Damien De Vienne / LBBE / CNRS Images)
Pour entraîner ces réseaux de neurones à fournir des réponses pertinentes, on utilise des simulations qui, pour une séquence donnée dans le génome, calculent les changements qu’elle peut subir au cours du temps. Cette évolution n’est pas uniforme dans la séquence. Certaines portions de la séquence ont plus de chances de changer que d’autres, et certaines parties changeront plus vite que d’autres. On peut ainsi simuler la manière dont ces changements suivent l’apparition de nouvelles espèces.
À partir de ces résultats (validés par les échantillonnages génétiques d’espèces ou de souches existantes), les modèles de réseaux de neurones seront entraînés à remonter le cours du temps. Partant du résultat final, ils pourront reconstituer l’arbre phylogénétique le plus fidèlement et précisément possible.
Des applications dans nombre de domaines liés à la biologie
Outre son application dans le domaine de la phylogénie et de l’étude de l’histoire de l’évolution des espèces sur la Terre, on trouve une autre application des techniques présentées en épidémiologie, où l’on étudie la propagation et l’évolution des maladies infectieuses afin de comprendre comment les virus, les bactéries et les autres agents pathogènes se propagent au sein d’une population au fil du temps. L’étude génomique se restreint alors à une seule espèce virale, sur une période bien plus courte que dans l’exemple précédent, mais l’application est similaire : prendre des échantillons de virus chez plusieurs personnes malades, les séquencer et reconstruire l’arbre phylogénétique construit sur ce virus et ses différents variants tout au long de l’épidémie pour faire de l’inférence, afin d’en extraire des informations pertinentes (vitesse de propagation, durée de l’infection, etc.).
Bien sûr, les calculs développés pour l’étude des génomes grâce à l’intelligence artificielle s’appuient sur des hypothèses simplificatrices qui, comme tout modèle, ne représentent le réel que dans une certaine mesure.
Le travail se poursuit pour améliorer la qualité et la fiabilité de ces prédictions phylogénétiques et, ainsi, permettre à la génomique de toujours mieux comprendre l’évolution du vivant.
_________________________
Ces recherches ont été financées en tout ou partie, par l’Agence nationale de la recherche (ANR) au titre du projet ANR-PIECES-AAPG2020. Cette communication est réalisée et financée dans le cadre de l’appel à projet Sciences Avec et Pour la Société - Culture Scientifique Technique et Industrielle pour les projets JCJC et PRC des appels à projets génériques 2020 (SAPS-CSTI-JCJC et PRC AAPG 20).



