Vous êtes ici
Le fabuleux destin de la cellule
Cet article a été publié dans le n°9 de la revue Carnets de science
C’est une opportunité exceptionnelle. Celle de réunir les technologies et les connaissances européennes autour d’un objet unique : la cellule. Le but ? Étudier les différentes étapes de sa vie, de sa forme saine jusqu’à sa trajectoire pathologique. « L’idée est de joindre les forces de toute la communauté scientifique, en s’appuyant sur trois grands piliers : l’étude des composants cellulaires, la réalisation de modèles pathologiques et l’intelligence artificielle », explique Geneviève Almouzni, directrice de recherche CNRS au sein du laboratoire Dynamique du noyau1. C’est ainsi qu’en 2019 est né le consortium européen LifeTime, cocoordonné par la chercheuse. Il réunit plus de 100 institutions de recherche et hôpitaux de 18 pays européens, dont le CNRS pour la France, et il a reçu le soutien des biotechnologies et de l’industrie pharmaceutique.
Des changements de trajectoires
Si les 40 000 milliards de cellules qui composent notre corps intéressent tant les chercheurs, c’est parce que leur vie n’a rien d’un long fleuve tranquille… De la naissance jusqu’à leur spécialisation, pour devenir des cellules du foie, de la peau ou du cerveau par exemple, ces « briques élémentaires » changent de morphologie et de fonctions (c’est la différenciation cellulaire). Une vie de changements perpétuels dans l’organisme selon des trajectoires qui, lorsqu’elles dévient de leur chemin, peuvent entraîner des maladies : cancer, maladie d’Alzheimer, maladies cardiovasculaires…
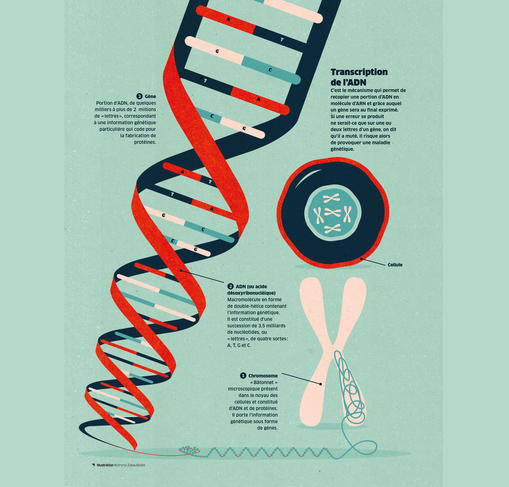
« Nous connaissons encore mal le fonctionnement complet de la cellule. L’objectif est de comprendre pourquoi une cellule s’engage dans une voie plutôt qu’une autre », indique Geneviève Almouzni. « Et ce en étudiant en profondeur les mécanismes intimes de leur vie », ajoute Giacomo Cavalli, responsable du consortium pour le CNRS et directeur de recherche à l’Institut de génétique humaine2. Au cœur de ces mécanismes notamment, l’expression génétique, cet ensemble de processus par lesquels l’information héréditaire, codée dans l’ADNFermer Ou acide désoxyribonucléique. Macromolécule en forme de double-hélice contenant l’information génétique. Il est constitué d’une succession de 3,5 milliards de nucléotides, ou « lettres », de quatre sortes : A, T, G et C. des noyaux des cellules, est lue pour aboutir à la fabrication de molécules dans l’organisme, par exemple des protéines. Ce sont aussi les interactions entre cellules et l’influence de l’environnement sur celles-ci qui doivent être étudiées de près.
En cartographiant le devenir des cellules neuronales dans le cadre de la maladie d’Alzheimer, par exemple, « nous pourrions à l’avenir être en mesure de détecter un signal avant que la maladie se développe », espère Xosé Fernández, directeur des datas à l’Institut Curie. Le jeu en vaut la chandelle : « Lorsque les patients souffrant de cette maladie arrivent en consultation, les cellules de leur cerveau sont déjà tellement dégradées que cela correspond presque à un stade métastasé de cancer », souligne le chercheur. Et même lorsque l’apparition de symptômes permet un diagnostic précoce de maladies chroniques, on ignore pourquoi l’état du tissu est souvent si largement dégradé.
À l’ère du séquençage haut débit
Si LifeTime naît aujourd’hui, c’est que les techniques d’analyse génomique et cellulaire sont en plein boom. Il y a trente ans, le projet génomeFermerEnsemble du matériel génétique d’une espèce. humain avait pour ambition de séquencer l’intégralité de nos gènesFermerPortion d’ADN, de quelques milliers à plus de 2 millions de « lettres », correspondant à une information génétique particulière qui code pour la fabrication de protéines. en une dizaine d’années. Aujourd’hui, grâce aux nouvelles méthodes de séquençage haut débit, cette opération est possible en moins d’une journée. « À l’époque nous pensions que le génome humain était composé de 50 000 gènes, alors qu’on sait aujourd’hui qu’il n’y en a que 20 000, ajoute Xosé Fernández. Et, grâce au séquençage haut débit, nous connaissons désormais le génome humain de référence », sorte de génome humain « moyen », mélange du séquençage de plusieurs personnes ne reflétant pas les diversités individuelles.

Même issues d’une seule personne, les données génétiques restent une moyenne issue d’un broyage de plusieurs cellules tout à fait différentes. Car chaque cellule, même si elle renferme le même code génétique de départ que les autres, aura exprimé en son sein un set de protéines différentes. La faute aux aléas de l’expression génétique et de nombreux autres facteurs, dont certains inconnus, aboutissant à une lecture différente de l’ADN, activant certains gènes et d’autres non...
C’est de ce constat que sont nées les techniques de séquençage à cellule unique « qui permettent d’extraire et d’analyser le génome d’une seule cellule, et confèrent une caractérisation individuelle de chacune », explique Xosé Fernández. Il est donc désormais possible d’étudier, cellule par cellule, les gènes exprimés ou les protéines produites. Et connaître ainsi la différence de dynamique entre une cellule cancéreuse (suite à une mutationFermerModification aléatoire de l’information génétique sur un petit nombre de nucléotides, ou « lettres », d’une séquence d’ADN lors de sa réplication ou « copie » en ARN. Une erreur, même sur une ou deux « lettres » seulement d’un gène, risque de provoquer une maladie génétique. génétique) et une cellule normale par exemple. « C’est ça le cœur de LifeTime », souligne Xosé Fernández.
Viennent aussi s’ajouter les progrès de la microscopie. « Avant les années 2000, la microscopie à fluorescence avait une résolution limitée à 250 nanomètres3. Or la structure de l’ADN, en double hélice, a une largeur de seulement 2 nanomètres… », souligne Giacomo Cavalli. Protéines, ARN ou complexes oncogéniquesFermerSéquence génétique qui, une fois transformée par un virus ou un autre agent, entraîne la formation d’une cellule cancéreuse. mesurent également entre 1 et 10 nanomètres. Avec ces anciennes techniques, il était donc impossible de visualiser les complexes des protéines ou les structures des acides nucléiques de taille inférieure à la limite de résolution. Mais depuis une dizaine d’années donc, la résolution des microscopes s’est considérablement améliorée, permettant de distinguer des structures biologiques de quelques nanomètres.
Une carte d’identité de la cellule
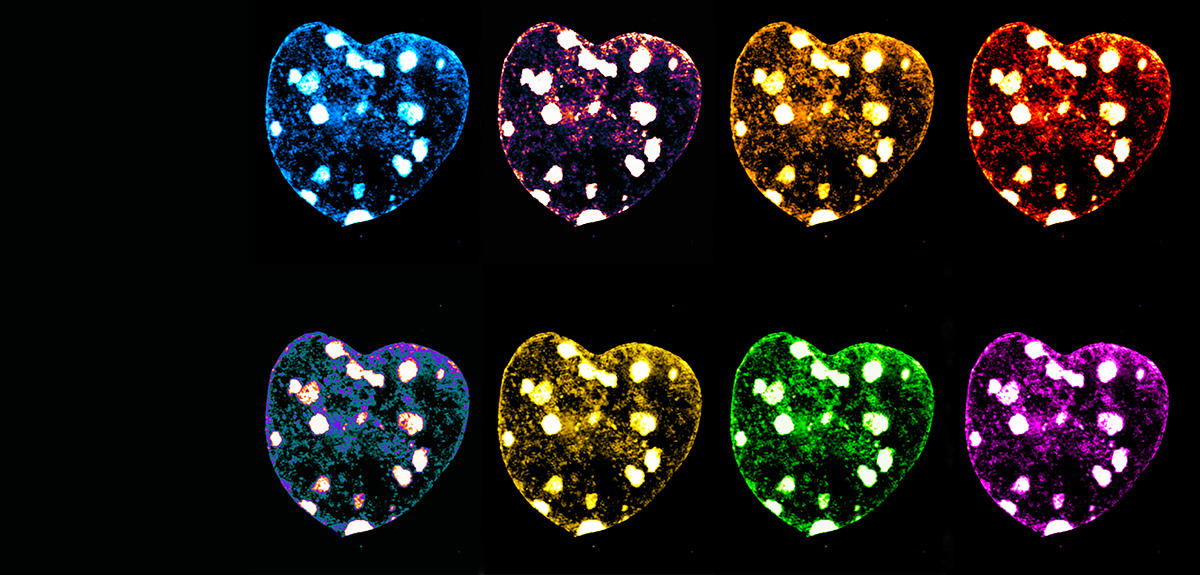
Et ce n’est pas tout : de nouvelles techniques de fluorescence de pointe sont aussi arrivées à maturation, comme le précise Marcelo Nollmann Martinez, directeur de recherche au Centre de biologie structurale4. « Avant, nous étions limités en termes de couleur, c’est-à-dire que nous n’avions que quatre ou cinq couleurs pour marquer les objets présents dans la cellule. C’était par exemple très difficile de visualiser la structure d’un chromosomeFermer« Bâtonnet » microscopique présent dans le noyau des cellules et constitué d’ADN et de protéines. Il porte l’information génétique sous forme de gènes. , avec seulement quatre points. » Désormais, la combinaison de technologies de microfluidique (art de manipuler les fluides aux échelles micrométriques) à la microscopie de fluorescence permet de superposer les signaux pour visualiser des dizaines de couleurs en même temps.
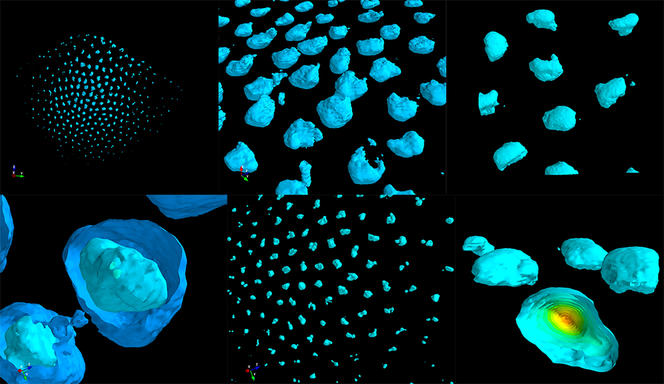

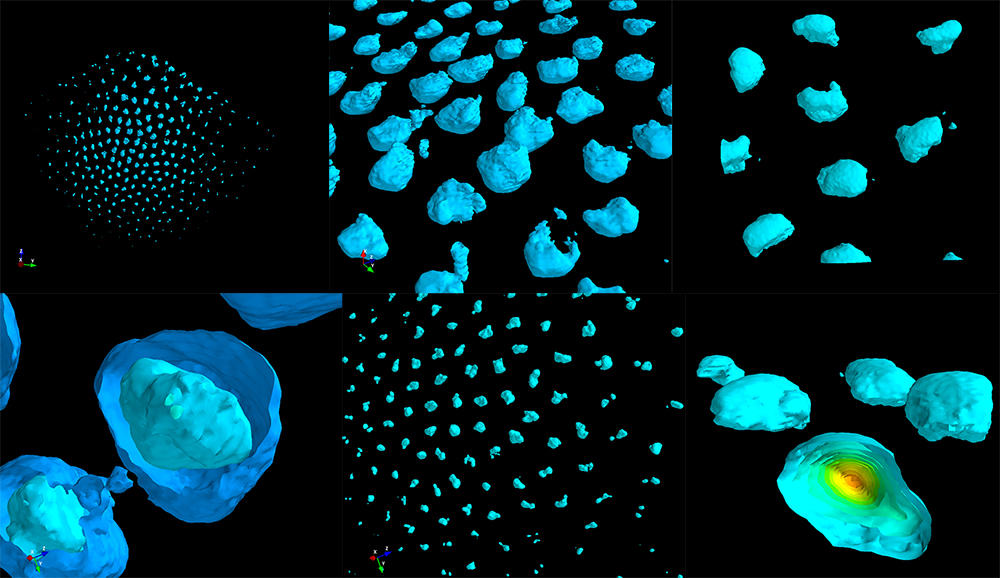
« Cela nous permet de visualiser simultanément et dans une même cellule 50 à 500 sites sur l’ADN ainsi que des centaines d’espèces d’ARN. Grâce à cette nouvelle méthode, nous pouvons maintenant retracer le parcours de l’ADN dans chaque cellule avec une résolution nanométrique », s’enthousiasme Marcelo Nollmann. Aujourd’hui, les chercheurs sont donc capables de détecter quels gènes sont exprimés par une cellule individuelle et comment leur expression est contrôlée par l’architecture tridimensionnelle du matériel génétique (on parle de structure 3D du génome). « Ces deux éléments nous permettent d’élaborer une carte d’identité de la cellule pour savoir si elle est normale ou pathologique », précise le chercheur.
Marcelo Nollmann et son équipe développent et utilisent ces nouvelles méthodes d’imagerie. Le but est d’étudier les changements de structure 3D du génome et du programme transcriptionnelFermerProcessus orchestrant, pour chaque cellule, la régulation de la transcription de milliers de segments d’ADN du génome afin de produire certains types de molécules d’ARN nécessaires au maintien de l’identité et des fonctions biologiques de la cellule. des différentes cellules pendant l’évolution d’une maladie comme le diabète. « Avant, nous prenions un pancréas en entier, broyé, et analysions la moyenne de l’expression génétique de toutes les cellules. » Or, le pancréas est composé de différents types cellulaires : les cellules bêta, qui produisent l’insuline, ou les cellules alpha, qui produisent du glucagon.
« Grâce aux nouvelles techniques de microscopie, on commence à comprendre comment les différents types cellulaires à l’intérieur d’un tissu ou d’un organe sont organisés spatialement et de quelle façon ils changent pendant l’avancement d’une maladie. En particulier, la visualisation de la structure du génome dans chaque type cellulaire nous permettra de comprendre les mécanismes de régulation d’expression génique en jeu pendant l’évolution de la maladie », explique Marcelo Nollmann.
Des modèles pour toutes les maladies
Tout l’enjeu est là : cartographier le parcours des cellules, leur changement d’expression génétique ou protéique, pour proposer un modèle applicable à toutes les maladies et utilisable par les laboratoires de recherche européens. Mutualiser les connaissances donc, les technologies, mais aussi les échantillons biologiques. Car certains pays possèdent des banques biologiques bien plus importantes que les nôtres. « En Israël par exemple, la population fait traditionnellement beaucoup de prises de sang. Ils possèdent donc une très grande base de suivi biochimique, sur plusieurs années, qui pourrait nous servir pour établir des modèles de cellules non pathologiques », imagine Xosé Fernández. Les données récoltées aux quatre coins de l’Europe « seront mises en commun à toute la communauté du consortium, pour que personne ne travaille dans son coin, et que la communauté scientifique gagne du temps en partageant les méthodes les plus efficaces », prévoit Geneviève Almouzni.
L’approche de suivi longitudinal de cellule unique sera complétée par des études sur organoïdes. Ces organoïdes sont des cultures en 3D de cellules qui miment le fonctionnement d’un organe. Certains sont déjà utilisés pour mimer un cancer du côlon par exemple. L’idée est de prélever des cellules de côlon au patient, puis de les dé-différencier, c’est-à-dire les remettre à l’état de cellule soucheFermerCellule indifférenciée, notamment capable de générer des cellules spécialisées (du foie, du cœur, de la peau, etc.) par différenciation.. « Ensuite, nous sommes capables de les re-différencier, pour créer finalement un mini-intestin que l’on pourra étudier », précise Geneviève Almouzni.


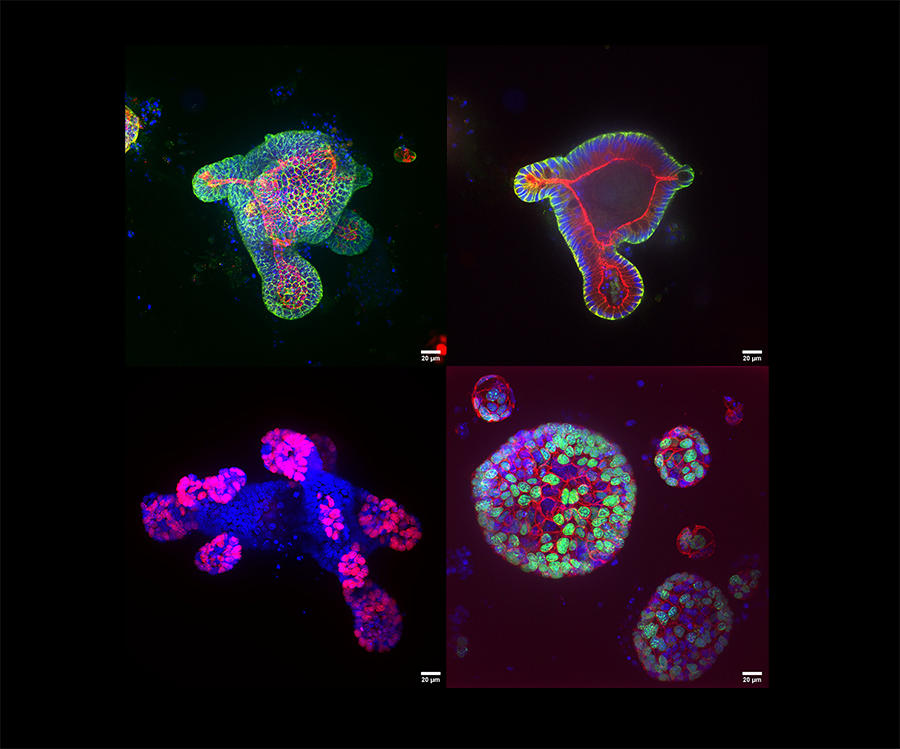
Le développement de ces organoïdes ouvre d’immenses espoirs pour tous les laboratoires qui pourront y avoir accès. Notamment celui de pouvoir, en culture, tester l’efficacité de nombreux traitements « ou l’influence d’agents pathogènes, pour comprendre comment ils altèrent ces organoïdes ou aboutir à la formation de cancers », commente Geneviève Almouzni. L’objectif est ainsi de mettre en commun une batterie de tissus biologiques « sur lesquels nous pourrons criblerFermerEn pharmacologie ou en génomique, techniques visant à étudier et à identifier des molécules aux propriétés nouvelles, biologiquement actives, parmi celles de banques de données. des milliers de molécules et regarder si elles sont efficaces », espère Giacomo Cavalli.
Des traitements mieux ciblés ?
Giacomo Cavalli, pour sa part, met également en avant des applications en neurobiologie. Alors que beaucoup d’études génomiques ont été réalisées sur des pathologies avec dysfonction cognitive comme l’autisme, on ne comprend pas toujours le rôle des gènes impliqués dans ces maladies. « En prélevant des cellules dans le sang de patients atteints de troubles neurodéveloppementaux, on peut les déprogrammer, les remettre à l’état de cellule souche, puis les reprogrammer pour qu’elles deviennent des neurones. » Les chercheurs disposent donc ainsi, sans prélèvement dans le cerveau, de neurones contenant le code génétique du patient qu’ils peuvent étudier à l’échelle de la cellule unique. « Nous nous sommes alors rendu compte que chez ces patients, certaines fonctions neurologiques étaient perturbées. Cette observation était impossible en se limitant à l’analyse des gènes, assure Giacomo Cavalli. Cela dégage de nouvelles pistes explicatives mais aussi thérapeutiques. »
Zoomer en détail sur la machinerie d’une cellule permettra aussi, espèrent les membres du consortium européen, de comprendre pourquoi certaines cellules tumorales répondent aux traitements et d’autres non. Les chercheurs ont d’ores et déjà à disposition des outils pour mimer les dysfonctions métaboliques et le processus tumoral. « Nous pouvons appliquer ces outils pour comprendre l’impact du cancer sur la cellule et peut-être intervenir le plus tôt possible », suggère-t-il.
La prévention des maladies cardiovasculaires est aussi en ligne de mire. Après un infarctus en effet, l’analyse de l’ARN des cellules cardiaques pourrait permettre de dégager des signaux moléculaires de guérison des séquelles de la crise cardiaque. Ou au contraire d’aggravation !
LifeTime a également pour vocation d’impliquer l’industrie pharmaceutique et de lui apporter des clés de compréhension. Un travail collaboratif qui permettra un partage de connaissances et d’outils, et contribuera à un développement plus efficace de solutions thérapeutiques personnalisées. Car, pour découvrir des traitements, la recherche pharmaceutique doit souvent tester des milliers de molécules et observer lesquelles fonctionnent ou non, « sans vraiment comprendre pourquoi », commente Giacomo Cavalli.
l’intelligence artificielle à la rescousse
ADN, protéine, ARN, métabolisme, environnement cellulaire… De toutes ces analyses émergeront une colossale quantité de données. L’analyse génétique de seulement 10 000 des cellules du corps humain « nécessite déjà une puissance de calcul considérable », signale Xosé Fernández. D’où l’intervention du troisième grand pilier de LifeTime : l’intelligence artificielle. Alors que les spécialistes des sciences du vivant récolteront les données, les bioinformaticiens s’occuperont de les traiter pour faire ressortir, par exemple, des variants génétiques ou des signaux précoces de maladie. « En obtenant une cartographie moléculaire du parcours d’une maladie, l’intelligence artificielle pourrait faire émerger un modèle de détection », espère Xosé Fernández.
Autre exemple, avec l’utilisation de l’historique médical des patients, comme l’explique Xosé Fernández : « Le cancer est généralement une maladie liée au vieillissement. L’âge moyen de nos patients est de 66 ans et ils ont souvent pris des traitements toute leur vie pour des maladies associées. Le machine learningFermerAussi appelé apprentissage machine. Technique d’intelligence artificielle où la machine apprend à partir des données, en l’absence de règles logiques dictées par l’humain programmeur. permet de croiser ces données médicamenteuses pour savoir si les traitements passés peuvent potentialiser, ou au contraire annihiler le traitement anticancéreux. »
LifeTime s’inscrira dans une approche éthique et sociétale de la science, combinant les aspects technologiques, fondamentaux, industriels et citoyens. « Nous mettons une importance cruciale sur la protection des données personnelles », souligne Geneviève Almouzni. « C’est tout l’intérêt d’un consortium européen », renchérit Xosé Fernández, car l’Europe a mis en place le Règlement général sur la protection des données (RGPD) en 2016, contrairement aux États-Unis par exemple. « L’objectif est de faire naître un système européen fédéré des données, à l’instar du Health Data Hub (plateforme des données de santé) créée en 2019 en France. »
La prochaine étape est désormais de décrocher des financements européens pérennes. En septembre 2020, les membres de LifeTime ont publié un article dans la revue scientifique Nature5 pour exposer les grandes perspectives du projet. Un article dont la signature « n’a pas pu rendre crédit à tous les chercheurs qui y ont contribué ! », regrette Geneviève Almouzni, soulignant l’importance de l’effort collectif dans cette recherche déterminante. « Nous sommes à un moment clé de l’étude de la cellule, c’est un rêve qui prend forme », s’enthousiasme la chercheuse. ♦
Pour en savoir plus
Le site du consortium européen LifeTime (en anglais) : https://lifetime-initiative.eu/
- 1. Unité CNRS/Institut Curie/Sorbonne Université.
- 2. Unité CNRS/Université de Montpellier.
- 3. Un nanomètre est égal à un millionième de millimètre.
- 4. Unité CNRS/Inserm/Université de Montpellier.
- 5. « LifeTime and improving European healthcare through cell-based interceptive medicine », N. Rajewsky, G. Almouzni, G., Gorski et al., Nature, septembre 2020.
Voir aussi
Auteur
Léa Galanopoulo est journaliste scientifique indépendante.