Vous êtes ici
C’est une véritable course contre la montre. Mais une course qui s’apparente davantage à un marathon qu’à un sprint. Face à la crise sanitaire, les scientifiques du Centre International de Recherche en Infectiologie de Lyon sont mobilisés pour trouver les molécules qui permettent de lutter efficacement contre le virus SARS-CoV-2. Impossible cependant de découvrir une nouvelle molécule dans l'urgence.
ITV
Le développement d'une nouvelle molécule, aujourd'hui c'est considéré comme un développement long, de 10 à 12 ans, avec des coûts qui sont très importants, de l’ordre du milliard de dollars.
Les scientifiques ne disposent pas de ces délais. Mais il existe une autre piste :
ITV Manuel ROSA CALAVATRA, Virologue
Le repositionnement de médicaments c'est le principe de réutiliser des médicaments qui sont déjà sur le marché pour de nouvelles indications thérapeutiques. C'est plutôt un temps de développement entre 5 et 6 ans et un coût considérablement réduit, aux alentours de 300 millions de dollars.
5 à 6 ans, c’est encore trop long. Pour réduire davantage le délai, l'équipe ne va pas tester les milliers de molécules potentiellement efficaces. Les molécules seront d’abord identifiées avec un ordinateur. Cette approche permet de réduire le nombre de molécules candidates pour ensuite les tester seules, mais également combinées les unes aux autres.
ITV Olivier Terrier, virologue
Ambiance “On voit les quantités de quelques nanolitres qui sont déposées au fond des puits…”
On réalise ce qu'on appelle un criblage “in silico”. Ce criblage “in silico” en quoi consiste-t-il ? À partir des échantillons de patients, on va déterminer l'empreinte que laisse le virus dans la cellule lors de l'infection. Et à partir de cette empreinte, on va aller identifier des médicaments qui sont capables de contrecarrer l’infection.
Le criblage informatique a permis d’identifier quelques dizaines de molécules ayant un effet sur les cellules infectées. Elles passent alors à l’étape suivante. Direction le laboratoire P3, un environnement contrôlé et confiné qui permet de manipuler les virus en toute sécurité. Les molécules sont maintenant testées sur des cellules d’épithélium respiratoire humain - un modèle in vitro qui se rapproche de la complexité qui existe dans nos poumons.
ITV
C'est un modèle dont on dispose au laboratoire, qui est obtenu à partir de cellules de patients, et qui présente vraiment les mêmes propriétés que ce qu’on retrouve chez les patients. Donc ça permet de tester les molécules dans des conditions qui sont le plus proches de la vraie vie, avec une capacité de prédire leur efficacité chez les patients qui est plus importante.
L’équipe est mobilisée depuis fin janvier pour lutter contre le COVID-19. Les chercheurs ont réussi à isoler SARS cov 2 à partir d’échantillons prélevés sur les premiers patients infectés en France.
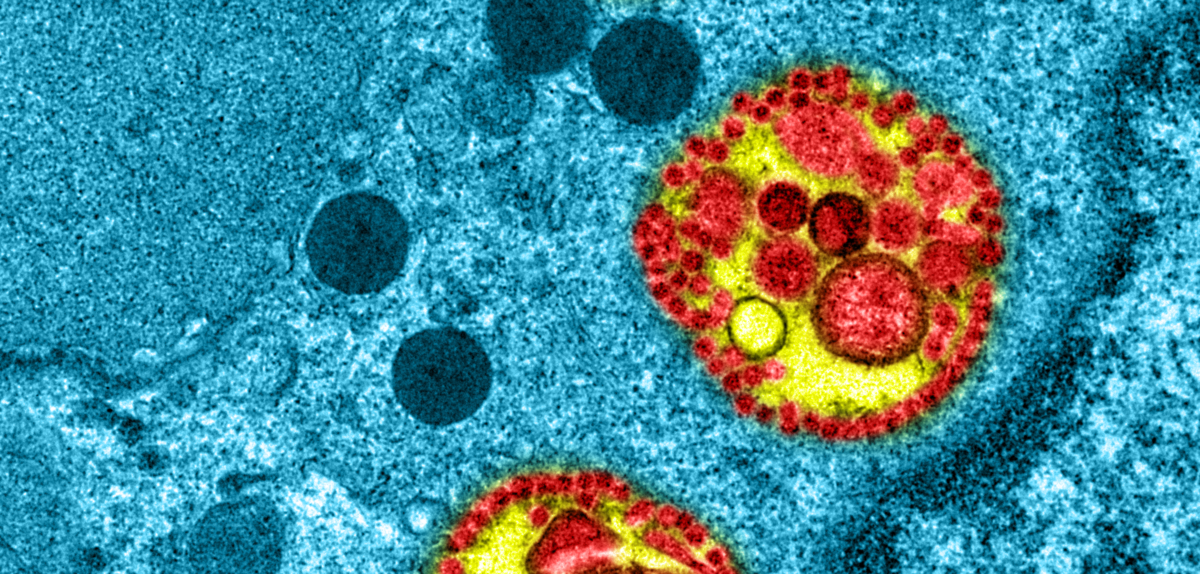
Ils ont pu ensuite produire les premières images de microscopie électronique du virus permettant de mieux comprendre la manière dont il se réplique dans l’épithélium respiratoire.
ITV.
À la surface de ces épithéliums, on voit très bien les cils. On voit ici des accumulations très importantes de matériel viral, de véritables usines à virus. Et puis ici, en petits chapelets, de nouveaux virus SARS-CoV-2 qui vont migrer et qui vont infecter de proche en proche les autres cellules de l'épithélium respiratoire.
Le travail de ce laboratoire a permis de valider au niveau pré-clinique plusieurs molécules qui font aujourd’hui l’objet d’évaluation chez des patients. Un effort au long cours, qui mêle travail appliqué et recherche fondamentale. Malgré les méthodes utilisées pour accélérer le processus, le temps de la recherche reste nécessairement long.
ITV Olivier Terrier
Le temps pour pouvoir aller de l’identification par ordinateur jusqu'à une validation “in vitro”, pour aller éventuellement chez des patients, ça peut aller de quelques semaines à quelques mois.
À ce jour on a évalué dans l'équipe plus de 80 molécules ou combinaisons de molécules jusqu'à l'étape d'épithélium, et parmi toutes ces molécules, on a bien évidemment testé les molécules dont tout le monde a entendu parler : le Remdesivir, le Lopinavir, l’Hydroxychloroquine, et bien évidemment on a testé bien d'autres molécules candidates.
Une des molécules qu’on a identifiée au labo, elle s’appelle le Diltiazem, c’est une molécule repositionnée, qui était initialement utilisée pour le traitement des angines de poitrine, et dont on a découvert une activité antivirale contre les virus de la grippe. En identifiant le mode d’action de cette molécule, on s’en rendu compte que cette molécule avait probablement un effet antiviral à large spectre, c'est-à-dire efficace contre beaucoup d'autres virus respiratoires. On a pu prouver rapidement, au début de l’épidémie, que c’était une molécule qui était efficace contre le SARS-CoV-2, “in vitro”. L'objectif de l'équipe aujourd'hui c’est de pousser cette molécule jusqu'à son évaluation clinique, seule ou en combinaison avec d'autres molécules antivirales.
Si le travail à accomplir reste conséquent, plusieurs pistes prometteuses se dessinent déjà dans cette quête du médicament contre SARS-Cov-2. Une course menée in vitro, in vivo… et aussi in silico.Quelles molécules contre le SARS-CoV-2 ?
Comment trouver un médicament efficace contre le virus SARS-CoV-2 en un temps record ? Une équipe de scientifiques du Centre International de Recherche en Infectiologie de Lyon (CIRI) cherche parmi les molécules déjà existantes. Une course contre la montre qui se déroule in vitro, in vivo et même... in silico.
Manuel Rosa-Calatrava (Inserm)
Centre International de Recherche en Infectiologie (CIRI)
CNRS / Inserm / ENS Lyon / Université Claude Bernard Lyon 1