Vous êtes ici
La bioinformatique, une alliée de taille contre le Covid-19

« La pandémie de Covid-19 a été un véritable test grandeur nature pour la bioinformatique. Elle a révélé combien les outils que nous développons sont désormais essentiels pour analyser les données et accélérer la recherche sur le virus ». Pour Hélène Touzet, du Centre de recherche en informatique, signal et automatique de Lille1, qui a coordonné un rapport récent sur le sujet (en anglais), le constat est clair : si les scientifiques ont pu percer en si peu de temps de nombreux secrets sur le SARS-CoV-2, de son identification à son fonctionnement en passant par sa propagation, c'est en grande partie grâce à la bioinformatique, ou biologie computationnelle. À la croisée des chemins entre l'informatique, les mathématiques, la biologie et la physique, cette discipline récente – une vingtaine d'années – fournit des modèles de calcul, des algorithmes et autres logiciels pour traiter l'avalanche de données biologiques et y découvrir des informations clés.
Séquencer le SARS-CoV-2
Dès le début de l'épidémie, la bioinformatique a montré toute son utilité. Après la détection en décembre 2019 des premiers malades infectés par le virus, les scientifiques chinois se sont lancés dans le séquençage du génome – le matériel génétique, constitué d'ARN, une longue chaîne de nucléotides – de ce nouveau pathogène à partir d'échantillons prélevés chez les patients. Et si les biologistes ont réalisé le séquençage en lui-même, ce sont des algorithmes de bioinformatique qui leur ont permis d'établir la séquence complète.

Les données brutes obtenues par séquençage sont en effet « illisibles » : elles sont composées de millions de petites séquences partielles, de quelques centaines de nucléotides, et d'origines diverses – des morceaux de génome du virus mais aussi du patient ou encore de bactéries. Pour y voir plus clair, un premier algorithme compare tous ces fragments à ceux présents dans une énorme base de données recensant tous les génomes du monde vivant connus à ce jour. Un peu à la manière d'une recherche Google sur Internet, on identifie alors toutes les séquences inconnues, appartenant donc au nouveau virus. Puis un second algorithme, fondé sur des méthodes d'optimisation de parcours dans des réseaux, remet tous les morceaux bout à bout dans le bon ordre.
Composé de quelque 30 000 nucléotides, le génome du SARS-CoV-2 est alors révélé, et ce, en un temps record : douze jours seulement ! « Cette rapidité s'explique à la fois par les progrès réalisés ces dix dernières années sur les algorithmes, mais aussi par l'accès en open source des logiciels et le partage des données génomiques à l'échelle mondiale. Cette tendance avait débuté dans les années 2010 avec l'épidémie d'Ebola notamment, mais elle est devenue la norme avec le Covid-19 », note Hélène Touzet.
Établir la généalogie du virus
Une fois le génome du virus établi, la bioinformatique a aussi permis d'en tirer de précieuses informations. La première concerne l'origine du virus. En comparant le nouveau génome à ceux des autres coronavirus connus, on a pu ainsi montrer que le SARS-CoV-2 présentait une plus grande similarité génétique avec des virus présents chez les chauves-souris qu'avec les virus humains du SRAS et du MERS, respectivement responsables d'épidémies en 2003 et 2012. Ce qui laisse fortement supposer que le SARS-CoV-2 nous vient à l'origine des chauves-souris.
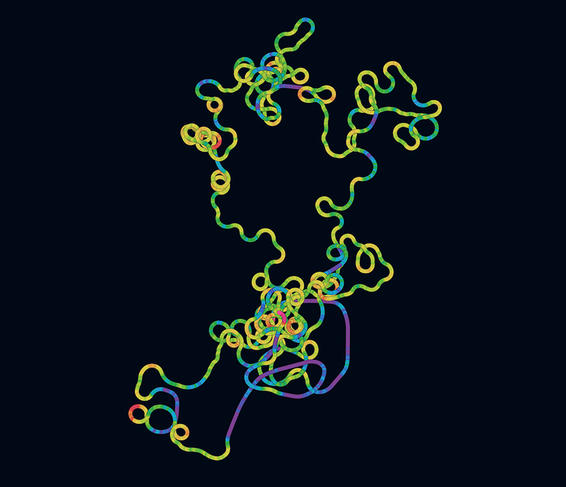

L'analyse comparative des génomes par des algorithmes a permis également de fournir aux scientifiques un premier aperçu de la biologie du virus. « Par analogie avec les autres virus dont on a déjà dressé le portrait-robot en laboratoire, nous avons pu identifier les séquences du génome codant pour des protéines, déterminer précisément quelles sont ces protéines, acide aminé par acide aminé, et ainsi pointer du doigt les points communs et les différences avec les protéines des autres virus », explique Hélène Touzet.
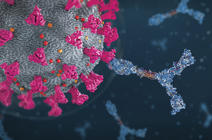
De cette manière, les biologistes ont pu découvrir les principales protéines nécessaires au fonctionnement du virus, en particulier celles impliquées dans l'infection des cellules humaines – comme tous les virus, SARS-CoV-2 a besoin d’une cellule hôte pour se multiplier et se propager. Parmi ces dernières, on trouve la fameuse protéine Spike, qui assure la reconnaissance des récepteurs cellulaires avant de pénétrer dans son hôte. Celle-ci est aujourd'hui au centre de l'attention des biologistes car c'est elle qui sert de cible aux vaccins contre le Covid-19 actuellement sur le marché.
Modéliser les mécanismes moléculaires
Même si elles sont d'une importance cruciale, les données génomiques ne suffisent pas à elles seules à comprendre le fonctionnement du virus une fois qu'il a pénétré dans le corps humain. Pour pouvoir le combattre, les biologistes doivent être capables de décrire en détail les mécanismes moléculaires en jeu. C'est là qu'intervient un domaine particulier de la bioinformatique – la bioinformatique dite « structurale » : grâce à des simulations numériques modélisant toutes les forces en jeu à l'échelle atomique, on tente de déterminer la structure tridimensionnelle des molécules et de comprendre comment leur structure influence ensuite la façon dont celles-ci interagissent entre elles.
Dans le cas du SARS-CoV-2, de tels modèles sont venus en renfort des expériences (cristallographie, cryomicroscopie électronique...) pour déterminer la structure de la protéine Spike et mieux comprendre sa dynamique lorsqu'elle s'accroche à une cellule hôte. Une étape clé pour la mise au point d’un vaccin.
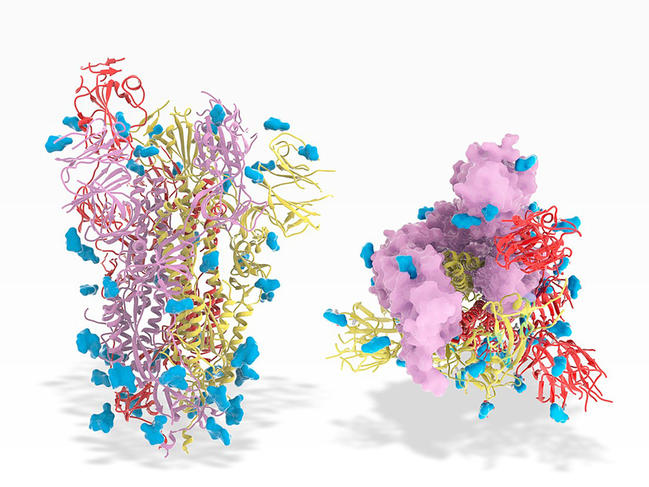
Ces modélisations structurales peuvent également permettre de prédire comment une mutation, c'est-à-dire une modification de la séquence d'ARN du virus qui se produit de manière aléatoire lorsque celui-ci se réplique, peut changer la forme d'une protéine – en remplaçant un acide aminé par un autre, par exemple – et ainsi sa fonction. Pour le SARS-CoV-2, les biologistes gardent donc un œil attentif sur les mutations qui pourraient affecter la protéine Spike, au point de rendre inefficace la stratégie vaccinale. « Une ambition ultime de la bioinformatique serait de pouvoir prédire la structure d'une protéine simplement à partir de la séquence du génome. Il y a encore du travail pour y parvenir, mais avec les outils actuels, on peut déjà estimer l'impact potentiel de mutations sur la conformation d'une protéine, et donc le risque de résistance aux vaccins », confie Hélène Touzet.
Suivre l’évolution du virus
L'intérêt de la bioinformatique ne s'arrête pas là. En plus de révéler l'origine du virus et sa biologie, les données génomiques permettent de retracer l'histoire de la propagation de l'épidémie. « L'accumulation régulière de mutations, au cours des infections, dans les génomes des virus – une à deux par mois en moyenne – peut être utilisée comme une ''horloge moléculaire'' : si des personnes sont infectées par des virus qui se ressemblent, cela signifie qu'elles sont proches dans la chaîne de transmission. Ainsi, en comparant les génomes des virus de patients infectés, et en utilisant les informations sur la date et le lieu d'échantillonnage, on peut retracer l'évolution de l'épidémie au cours du temps et de l'espace », explique Samuel Alizon, directeur de recherche CNRS au laboratoire Maladies infectieuses et vecteurs : écologie, génétique, évolution et contrôle2.
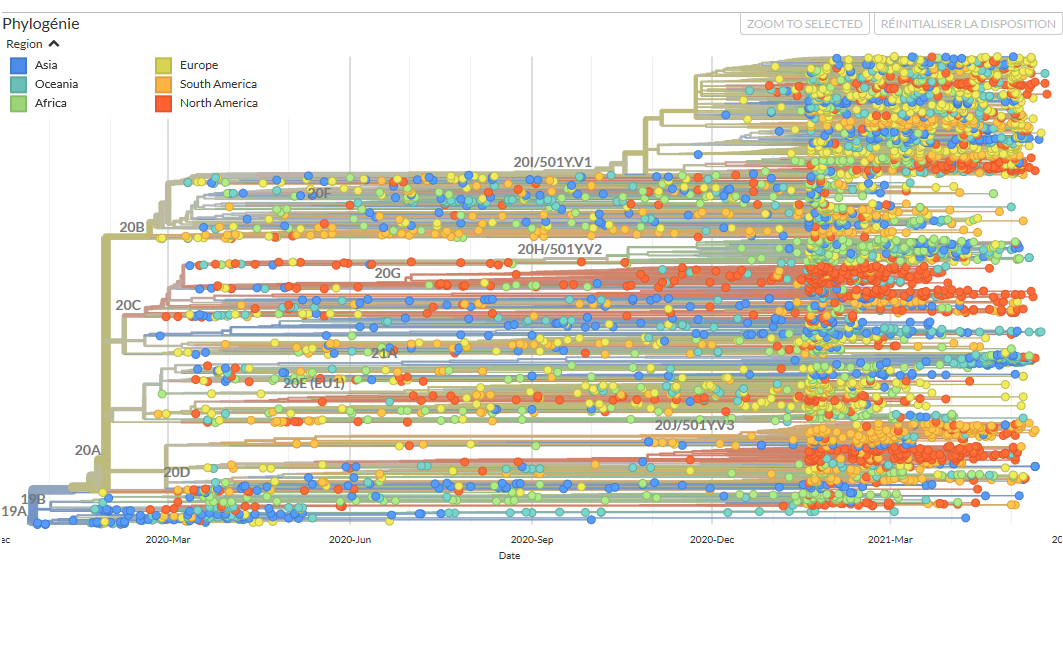
Ce jeune champ de recherche, la phylodynamique, est en plein essor avec le Covid-19. Concrètement, les bioinformaticiens utilisent les données de génomes pour construire un arbre phylogénétique, sorte d'arbre généalogique reliant les différentes infections dont les virus ont été séquencés. Ils utilisent pour cela le principe statistique du maximum de vraisemblance : l'arbre ainsi établi est celui qui explique le mieux les liens de parenté entre les séquences dont on dispose.
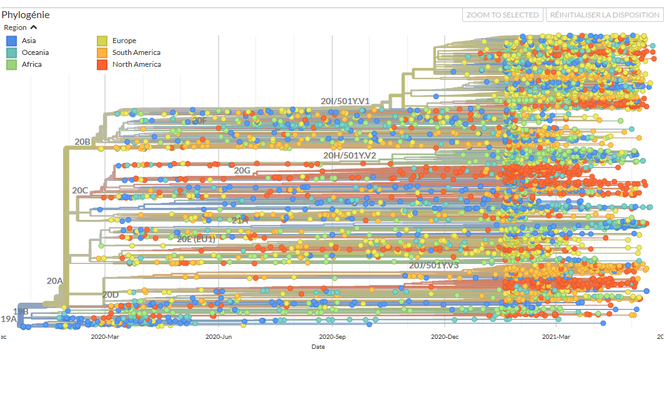
De ces arbres phylogénétiques peuvent être tirées énormément d'informations, notamment sur l'origine de l'épidémie. Ainsi, en comparant une cinquantaine de séquences virales recueillies au début de l'épidémie chez des patients chinois, les scientifiques ont observé une faible diversité génétique du virus, suggérant que celui-ci était apparu très récemment – entre août et décembre 2019 – dans la population humaine, à la suite d'une seule contamination par un animal, et qu’il n’y a pas eu ensuite d’autres transmissions de l’animal à l’humain.
Élucider les origines de l’épidémie
Ces arbres généalogiques permettent aussi d'éclairer l'histoire de l'épidémie, pays par pays. En France, par exemple, l'analyse par des chercheurs, parmi lesquels Samuel Alizon, de toutes les séquences génomiques disponibles en mars 2020, a révélé qu'elles descendaient toutes d'un ancêtre commun datant de l'apparition de l'épidémie en Chine. Le fait que l'on trouve ainsi dans l'Hexagone plusieurs lignées virales issues de Chine laisse penser que plusieurs « importations » indépendantes du virus ont concouru à la première vague épidémique. Au Royaume-Uni, où l’on dispose de bien plus de séquences encore, ce nombre a pu être estimé à plus d'un millier.
Autre illustration, à l'échelle d'une ville cette fois : à Boston, des chercheurs américains ont montré qu'une conférence commerciale internationale, qui s'est déroulée en février 2020, a été à l'origine d'un événement de super-propagation qui aurait indirectement conduit, selon leurs estimations, à plus de 100 000 contaminations dans le monde au cours des neuf mois suivants. Un travail clé pour mieux comprendre les causes de tels événements et pouvoir les éviter à l'avenir.
« La phylodynamique est un outil extrêmement précieux pour suivre une épidémie, d'autant que les données qu'elle utilise ont l'avantage de minimiser à la fois certains biais d'analyse – par rapport aux données d'incidence (le nombre de nouveaux cas par unité de temps, Ndlr) issues du dépistage – et les risques éthiques – par rapport aux données de traçage numérique », avance Samuel Alizon. À l'avenir, son équipe voudrait même aller plus loin en combinant les données génomiques aux données épidémiologiques traditionnelles. Pour renforcer plus encore la surveillance du virus. ♦
Voir aussi
Auteur
Julien Bourdet, né en 1980, est journaliste scientifique indépendant. Il a notamment travaillé pour Le Figaro et pour le magazine d’astronomie Ciel et Espace. Il collabore également régulièrement avec le magazine La Recherche.