Vous êtes ici
Covid-19 : ces variants qui ont changé la donne
Temps de lecture : 16 minutes
Alors qu’on pouvait commencer à espérer une sortie de la crise sanitaire en quelques mois, l’apparition cet hiver de différents variantsFermerOrganisme qui se distingue du virus d’origine par une ou plusieurs mutations. Quand celles-ci modifient son impact (en termes de transmissibilité, de virulence ou d’échappement immunitaire), on parle de « variant d’intérêt ». du virus du Covid-19 l'a, au contraire, fait repartir de plus belle. Malgré ce timing délétère, l’émergence de mutations reste un phénomène naturel qui a d’autant plus d’occasions de survenir que l’épidémie se propage. Mais qu’est-ce qui distingue les variants apparus en Angleterre, en Afrique du Sud et au Brésil des toutes premières souches ?
« La terminologie est souvent utilisée de façon brouillonne, au risque de laisser passer des informations erronées », souligne Bruno Lina, professeur des universités, chercheur au Centre international de recherche en infectiologie1 et praticien hospitalier aux Hospices civils de Lyon, qui est également directeur du Centre national de référence des virus infectieux respiratoires et membre du conseil scientifique. « Le Covid-19 est provoqué par un virus unique : le SARS-CoV-2. Il évolue selon plusieurs lignées qui, après des modifications de leur génome (c’est-à-dire de leur matériel génétique, Ndlr), obtiennent parfois un avantage par rapport aux autres. »
Comme celui des autres coronavirus et contrairement à celui des êtres vivants, le génome du SARS-CoV-2 est entièrement constitué d’ARNFermerAcide ribonucléique, très proche chimiquement de l’ADN dont il est une sorte de copie, et portant de l’information génétique sous forme d’une longue suite de nucléotides (ou « lettres »).. Enchaînement de quatre nucléotides, adénine (A), guanine (G), cytosine (C) et uracile (U), cet ARN peut subir des mutationsFermerModification aléatoire de l’information génétique sur un, deux, ou un petit nombre de nucléotides (ou « lettres ») d’une séquence d’ADN ou d’ARN lors de sa réplication ou « copie ». Plus un virus se multiplie, plus ces mutations sont donc susceptibles d’apparaître., c’est-à-dire qu’un ou plusieurs de ses nucléotides peuvent être ajoutés, remplacés ou supprimés, et ce de manière aléatoire. Pour identifier ces variations, on leur attribue un chiffre qui les localise sur le génome et sur les protéines qui en résultent, ainsi que des lettres indiquant la nature des modifications.
Mutant et opportuniste
Si l’idée d’un virus mutant peut effrayer, ces modifications n’ont généralement pas d’impact sur sa propagation ou sa dangerosité. Il existe ainsi une lignée française qui ne s’est pas diffusée au reste de l’Europe. Au total, quelques dizaines de lignées existeraient à différentes échelles dans le monde. Les choses se compliquent quand les mutations modifient la transmissibilité ou la virulence du virus, ou encore sa sensibilité aux traitements et vaccins. « Dans le jargon des virologues, on ne parle de variant que lorsque les mutations touchent à la réponse antigénique du virus, poursuit Bruno Lina. Les anticorps qui réagissent avec le virus originel ne le font plus aussi bien, voire plus du tout, avec le variant. Ainsi, alors que la réponse immunitaire induite par le virus originel reste adaptée pour le “variant anglais”, elle semble l’être moins pour les variants brésilien et sud-africain. »
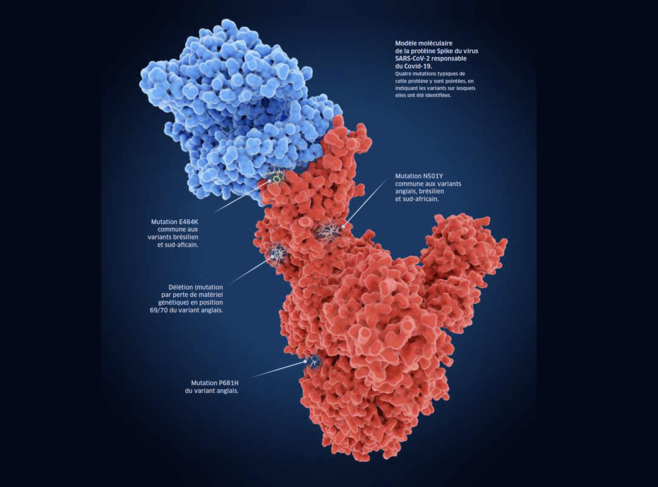
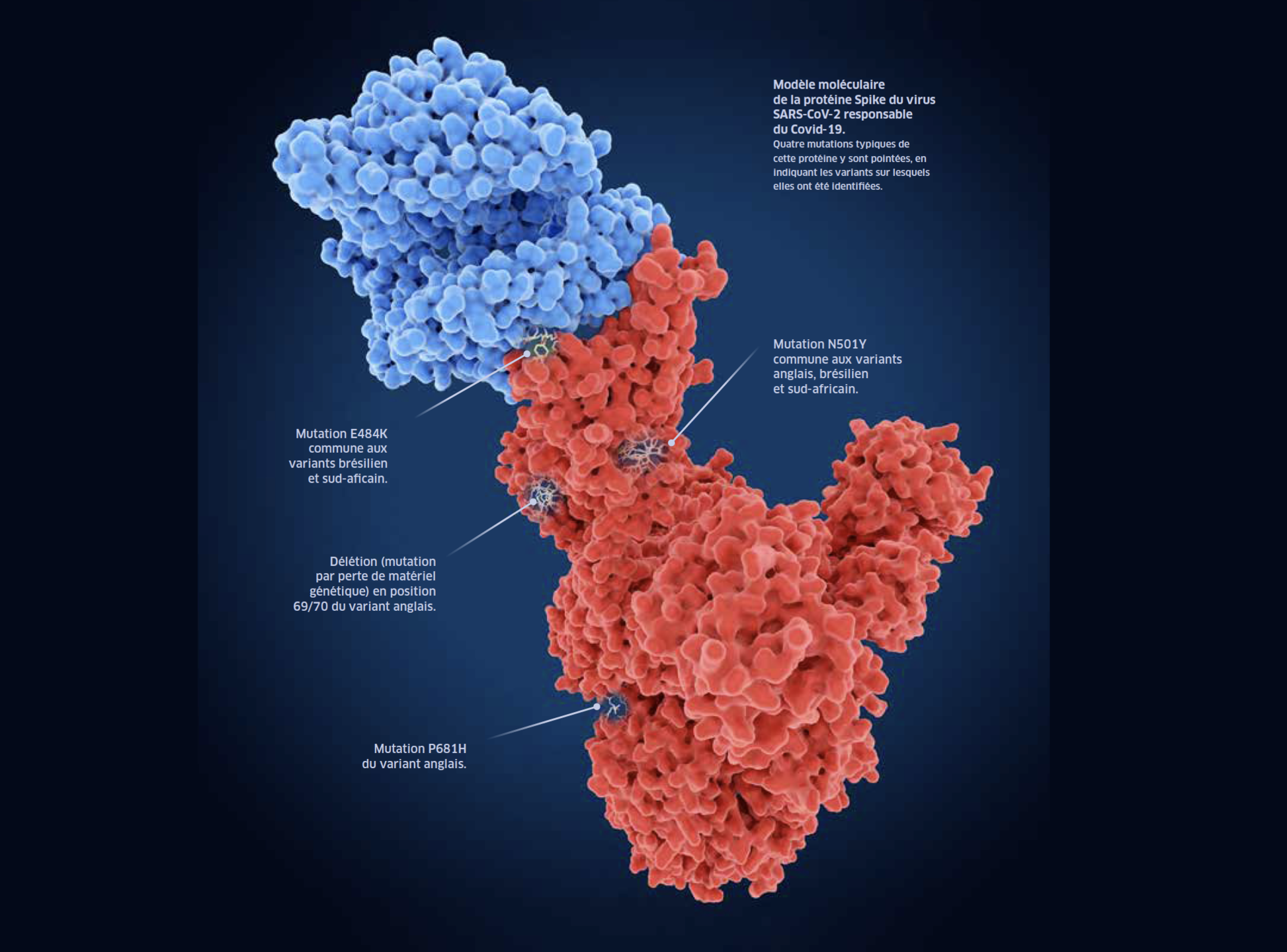
Le SARS-CoV-2, comme tous les virus, a besoin d’une cellule pour se multiplier et se propager. Il doit donc entrer dans sa cible pour détourner les machineries cellulaires à son profit. Dans le cas du SARS-CoV-2, la protéine SpikeFermerProtéine en forme de pique se trouvant sur la surface du virus du Covid-19 et qui lui permet de pénétrer dans les cellules à infecter. reconnaît les récepteurs cellulaires avant de pénétrer dans son hôte. La version britannique possède justement la mutation N501Y sur cette protéine, ce qui améliore l’entrée du virus dans la cellule et pourrait le rendre plus contagieux. Les génomes des variants d’Afrique du Sud et du Brésil contiennent aussi cette mutation, mais ils la cumulent avec la mutation E484K, qui neutralise une partie de la réponse immunitaire des personnes immunisées contre les lignées historiques de SARS-CoV-2. Des points communs qui ne sont pas forcément une si mauvaise nouvelle…
En effet, « si les mutations convergent ainsi vers les mêmes points chauds d’évolution, ce que la surveillance des lignées laisse présager, le virus pourrait finir par se trouver dans un véritable goulot d’étranglement : il n’aurait plus assez de marge de manœuvre pour produire de nouveaux variants et pour continuer de s’adapter aux humains, avance Bruno Lina. La situation pourrait alors finir par se stabiliser. » Un statu quo qui serait appréciable vu l’efficacité annoncée des vaccins déjà au point.
Moins optimiste, Samuel Alizon, directeur de recherche CNRS au laboratoire Maladies infectieuses et vecteurs : écologie, génétique, évolution et contrôle (Mivegec)2 et membre du groupe de modélisation de l’équipe Évolution théorique et expérimentale (ETE) du Mivegec, considère que « le champ des possibles a changé car les variants peuvent continuer à évoluer par mutation dans un paysage adaptatif qui nous est, pour le moment, inconnu ».
Filet de sécurité embarqué
Mais quelle est sa capacité de mutation et d’où vient-elle ? « Grippe, dengue, Zika, chikungunya, sida, Covid-19… toutes les pandémies virales de ces dernières années ont été causées par des virus à ARN, énumère Isabelle Imbert, professeure à Aix-Marseille Université et membre du laboratoire Architecture et fonction des macromolécules biologiques3. Ils s’adaptent plus facilement que les virus à ADN, car ils ne réparent pas les erreurs qui apparaissent lors de la réplication de leur génome et mutent ainsi plus souvent. Les coronavirus font cependant figure d’exception : ce sont les seuls représentants de cette famille à quand même posséder un mécanisme de correction. »

Les coronavirus se distinguent en effet par la longueur de leur génome : alors que le virus à ARN de l’hépatite C contient moins de 10 000 nucléotides, celui du SARS-CoV-2 en compte 30 000 ! Avec autant de possibilités de mutations, le virus disparaîtrait rapidement sans ce « filet de sécurité » correctif. Grâce à celui-ci, il n’évolue en moyenne que de deux mutations par mois, soit cent fois moins que le VIH. Ainsi, les génomes de la lignée anglaise n’ont en moyenne que dix-sept différences avec celui des souches historiques apparues il y a plus de quatorze mois. « Dans une cellule infectée, le génome du virus est répliqué grâce à une réaction chimique de polymérisationFermerRéaction chimique ou procédé permettant de concevoir de grosses molécules (contenant un grand nombre d’atomes) en faisant réagir entre elles de plus petites molécules. lors de laquelle les erreurs de copie peuvent apparaître, détaille Isabelle Imbert qui a publié plusieurs travaux sur le sujet. En cas de problème, la polymérisation est ralentie grâce à deux petites protéines virales permettant le recrutement de l’exonucléase, une enzyme elle aussi codée par le virus. Capable de couper l’ARN, celle-ci “enlève” le mauvais nucléotide incorporé par erreur. Ce système de correction, dit “proofreading”, existe aussi chez les organismes à ADN. Sans cette série de réactions, les coronavirus muteraient vingt fois plus vite. »
Fort taux de transmission
Dans le traitement par trithérapie contre le VIH, une des molécules vise justement la phase de polymérisation du génome du virus. La combinaison des médicaments permet de l’attaquer sur plusieurs fronts, l’empêchant de muter suffisamment vite pour se défendre contre les trois produits à la fois. Comme certaines activités enzymatiques virales impliquées ne sont pas naturellement présentes dans nos cellules, les effets secondaires sont réduits.
Dans le cas du SARS-CoV-2, le complexe de réplication est lui aussi une cible thérapeutique de choix, en plus de la protéine Spike sur laquelle se fondent la plupart des stratégies vaccinales. « De nombreux travaux scrutent la protéine Spike selon toutes ses mutations connues et mesurent la neutralisation des anticorps provenant de patients vaccinés, détaille Isabelle Imbert.
Dans ce suivi de l’évolution des variants, les vaccins dits à “ARN messager” ont l’avantage d’être très adaptables car il est facile de produire de nouvelles séquences d’ARN codant la protéine Spike avec ses mutations. Cette réactivité est moindre dans le cadre d’une stratégie vaccinale fondée sur l’injection de la protéine Spike purifiée (c’est-à-dire après l’avoir isolée du reste du virus, comme c’est le cas dans la stratégie vaccinale de Novavax, Ndlr). »



Selon le bilan du 11 mars de Santé publique France, sur les personnes testées positives, environ 66 % seraient infectées par la lignée britannique et 5 % par les variants brésilien et sud-africain (ces chiffres passent à 79,9 % pour le variant britannique et 4,4 % pour les variants brésilien et sud-africain selon le bilan du 1er avril, Ndlr). Les modèles épidémiques s’intéressent en particulier au nombre de reproductions qui désigne le nombre de personnes contaminées par chaque nouveau patient infecté. Noté R0 en début d’épidémie (car il correspond alors à une constante, valeur de référence), ce nombre peut ensuite varier au cours du temps (grâce aux gestes barrières par exemple) et est ensuite noté Rt. Quand il est inférieur à 1, l’épidémie recule. Rt était de l’ordre de 0,9 en France avant l’émergence des variants dans le pays, ce qui signifie que 10 % de personnes en moins étaient touchées à chaque cycle d’infection. Mais avec des souches de virus qui sont environ moitié plus contagieuses, Rt est repassé au-dessus de 1. Les mesures de protection doivent donc s’adapter à la situation qui reste malheureusement inédite.
La plupart des épidémies de l’histoire ont eu lieu alors que les chercheurs et les médecins ne disposaient pas d’outils génétiques pour retracer les lignées. Celles provoquées par des coronavirus, comme le SRAS (Syndrome respiratoire aigu sévère, apparu en 2002 en Chine) et le MERS (Coronavirus du syndrome respiratoire du Moyen-Orient, apparu en 2012 en Arabie saoudite), ont été rapidement contenues car elles présentaient peu de contaminations par des personnes asymptomatiques. L’isolement des malades suffisait à contrôler l’épidémie.
Modélisation virale
« Nous n’avons pas d’exemple bien connu d’apparition de variants de virus avec une transmission respiratoire aussi importante, regrette Bruno Lina. Nous pouvons en revanche penser au chikungunya qui ne posait pas de gros problèmes quand il n’était transporté que par une seule variété de moustiques. Une série de mutations lui a permis de passer dans davantage d’hôtes, dont des moustiques urbains, et à partir de là le virus s’est répandu d’un coup dans l’ouest de l’océan Indien. » Le développement de l’immunité a fini par avoir raison de l’épidémie car ces mutations n’ont pas concerné la réponse antigénique du virus du chikungunya. À l’inverse, les évolutions du virus de la grippe l’aident à réinfecter les personnes qui ont été atteintes précédemment. Entre ces deux cas de figure, il reste difficile de savoir comment SARS-CoV-2 va se comporter.
Les modèles épidémiologiques permettent d’y voir plus clair. « Nous avons commencé à travailler sur l’épidémie en mars 2020, quand nous avons compris que la proximité du virus avec celui du SRAS avait donné un faux sentiment de sécurité et que les premiers chiffres d’incidence avaient vraisemblablement été très sous-estimés », explique Mircea Sofonea, maître de conférences à l’université de Montpellier et membre du Mivegec.
Il dirige le groupe de modélisation de l’épidémie de Covid-19, fondé par l’équipe ETE. Les modèles améliorent les données issues du dépistage car les personnes testées ne sont pas forcément représentatives de la population globale. Tous les chiffres doivent ainsi être lissés et corrigés pour obtenir un état des lieux le plus juste possible sur l’épidémie.
« Nos outils aident à renseigner de façon quantitative, et non avec des intuitions ou des modèles verbaux », insiste Mircea Sofonea. Le groupe de travail a par exemple conçu les applications Rt2 et CovidIci, qui permettent de visualiser en temps réel la dynamique de l’épidémie. « En absence de mesures sanitaires, le R0 des souches historiques est de 3, mais il atteint 4 pour les trois variants qui font l’actualité », s’inquiète Mircea Sofonea. Ainsi, chaque personne infectée en contaminerait quatre autres, provoquant une propagation vertigineuse de l’épidémie. « Le dernier déconfinement en décembre 2020 s’est opéré alors que le niveau de circulation du virus restait élevé, et le couvre-feu de 18 heures généralisé à toute la France qui a suivi mi-janvier n’a permis que de figer cet état. Mais en tant que chercheurs, nous ne prescrivons pas de politique sanitaire, notre rôle est de produire des analyses quantitatives. »

En plus de sa répartition spatiale, le parcours du virus s’étudie aussi dans le temps. « La phylodynamique construit les arbres généalogiques des infections à partir du génome des virus, décrit Samuel Alizon. Le SARS-CoV-2 vient a priori des chauves-souris et serait passé chez l’être humain entre août et novembre 2019. À part l’acquisition de la mutation D614G, on n’a pas vu de propagation massive de mutations conférant un avantage au virus jusqu’à l’apparition des variants fin 2020. » Comment des lignées portant des mutations similaires ont-elles émergé de façon si rapprochée dans le temps et en plusieurs endroits différents ? Cela peut s’expliquer à l’image du phénomène d’épistasie, qui décrit comment des gènes interagissent les uns avec les autres. Certaines mutations, comme la N501Y présente dans les lignées apparues en Angleterre, en Afrique du Sud et au Brésil, n’auraient un impact majeur que combinées avec une série d’autres mutations, bénignes en l’absence des autres.
Réponse immunitaire contournée
« Si les risques qu’un virus isolé accumule toutes les mutations favorisant sa propagation sont statistiquement très faibles, ils ne sont plus négligeables lorsque plusieurs millions de personnes sont infectées, prévient Samuel Alizon. De plus, l’immunisation des populations modifie les pressions de sélection. » Le chercheur prend comme exemple la ville de Manaus, au Brésil, où les autorités ont laissé l’épidémie se propager.
Cette approche, en plus de provoquer une catastrophe sanitaire en 2020, a peut-être conduit à l’évolution d’un variant capable de recontaminer certains anciens malades malgré leur immunité à la souche historique, si chèrement acquise. Une autre hypothèse expliquant l’émergence des variants est celle de patients immunodéprimés, chez lesquels on sait que les virus peuvent se répliquer plusieurs mois grâce à une réponse immunitaire affaiblie de la personne. Concernant la virulence du virus, c’est-à-dire sa capacité à se multiplier rapidement, augmentant la charge virale du malade, elle-même susceptible de provoquer des formes graves de l’infection, on ne sait pas si elle a changé avec les variants.
Quant aux conséquences sur le nombre de décès, une étude publiée le 10 mars dans la revue British Medical Journal a montré que le variant anglais était 64 % plus létal sur les cas symptomatiques que les souches historiques. Et une modélisation, réalisée fin décembre 2020 et publiée le 3 mars dans Science, montrait que ce variant, bien plus contagieux que les lignées précédentes, pouvait se révéler plus mortel au niveau de la population car causant plus d’infections. « Avec le nombre de décès que nous avons connu, nous aurions déjà atteint l’immunité collective si le virus causant le Covid-19 était aussi virulent que celui de la grippe, mais il l’est dix fois plus, déplore Samuel Alizon. Nous n’avons à ce jour pas de données pour dire si les variants vont gagner en virulence, mais leur contagiosité élevée risque de causer une mortalité accrue. Les modèles épidémiologiques suggèrent qu’une fois la population immunisée, les épidémies causées par ce virus pourraient être bien moins mortelles et plus semblables aux coronavirus saisonniers qui nous infectent tous les hivers. Mais il est encore trop tôt pour savoir si cela se produirait et à quelle échéance, surtout avec l’inconnue de l’étendue de la capacité des variants à échapper à la réponse immunitaire tout en demeurant virulents. » ♦
Retrouvez tous nos articles et podcasts consacrés à l'épidémie dans notre dossier :
Covid-19: la recherche mobilisée
Voir aussi
Auteur
Diplômé de l’École supérieure de journalisme de Lille, Martin Koppe a notamment travaillé pour les Dossiers d’archéologie, Science et Vie Junior et La Recherche, ainsi que pour le site Maxisciences.com. Il est également diplômé en histoire de l’art, en archéométrie et en épistémologie.