Vous êtes ici
Covid-19 : l'indispensable apport de la science des données

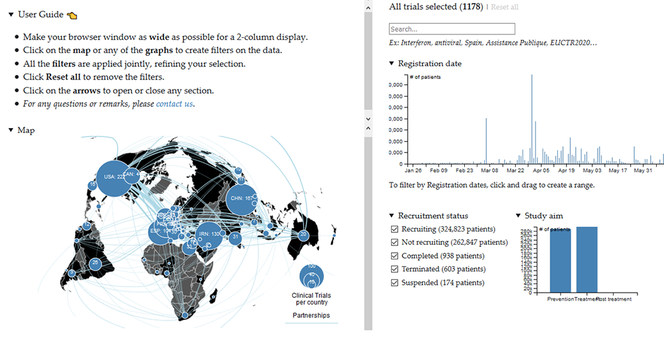
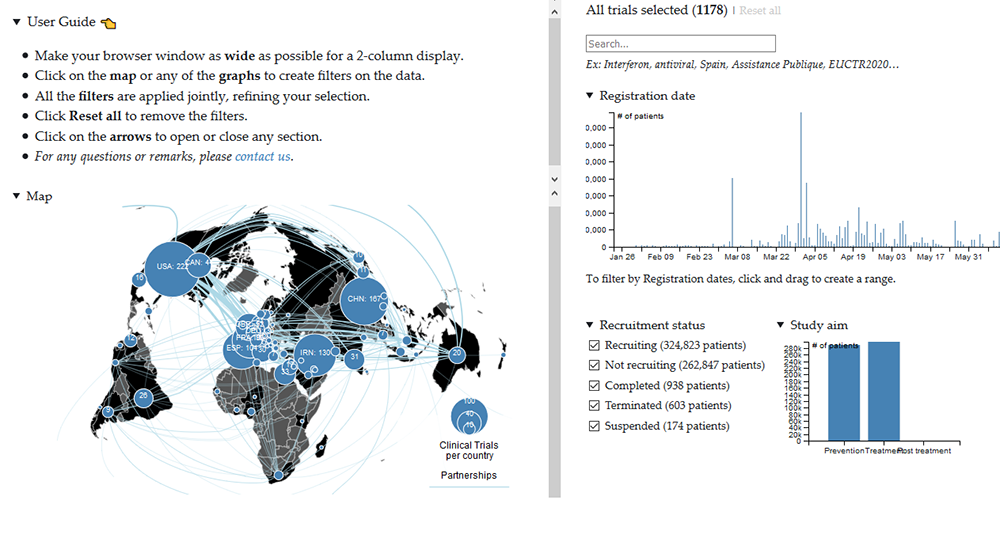
La recherche mondiale s’est mobilisée face à l’épidémie de Covid-19, mais difficile de se coordonner à l’échelle planétaire dans une telle urgence. Philippe Ravaud, directeur du Centre de recherche en épidémiologie et statistiques (Cress)1 et Isabelle Boutron, directrice de l’équipe Methods2 du Cress, ont monté un collectif international pour créer un site dédié : covid-nma.com « Alors que nous n’avons encore que peu de connaissances sur la maladie, médecins et responsables prennent tous les jours des décisions aux conséquences importantes sur la santé publique, déplore Isabelle Boutron. Devant l’urgence de la situation, nous voulons aider à la planification des études cliniques et donner accès aux informations nécessaires. » Covid-nma fournit une cartographie dynamique de toutes les études cliniques évaluant les méthodes de traitement et de prévention contre le Covid-19. Chaque jour, l’équipe collecte, explore, analyse et synthétise les données disponibles, puis en publie les résultats.
Un appel entre chercheurs
Mais la quantité d’information disponible dans les registres d’essais et dans les bases de données est considérable, et une extraction manuelle impliquerait des moyens humains beaucoup trop importants. Les chercheurs ont donc contacté le CNRS pour recevoir l’aide de ses spécialistes. Mokrane Bouzeghoub, directeur adjoint scientifique de l’Institut des sciences de l’information et de leurs interactions du CNRS et professeur à l’université de Versailles, a alors lancé un appel à volontaires qui a mobilisé des équipes de huit unités de recherche3, ainsi que le groupement de recherche Masses de données, informations et connaissances en sciences (MaDICS). Le travail accompli en deux mois, par plus d’une trentaine d’ingénieurs et chercheurs, a permis parallèlement d’extraire, intégrer et visualiser les données à partir de multiples sources et textes scientifiques.
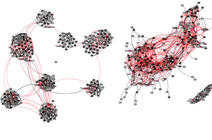
Sarah Cohen-Boulakia, directrice adjointe du Laboratoire de recherche en informatique4 et directrice de MaDICS, coordonne l’intégration et la collecte automatique des données issues des différents registres nationaux et internationaux d’essais cliniques. « Ces registres fournissent des informations similaires, mais ils ne sont remplis ni de la même manière ni avec la même précision, regrette Sarah Cohen-Boulakia. Certains sont faits pour être lus par des humains, d’autres pour être traités par des machines. » L’intégration de données vise à les présenter avec une apparente uniformité, alors qu’elles proviennent de sources disparates. Elle permet par exemple d’identifier l’utilisation d’une molécule dans plusieurs études cliniques, même si elle y est différemment décrite par sa formule chimique, son nom générique ou encore un de ses noms commerciaux. Des problèmes qui peuvent être facilement corrigés à la main sur de petits tableaux, mais en traiter des milliers réclame des procédures automatisées robustes afin d’en extraire les éléments pertinents.
Le principal terrain de chasse des équipes de Sarah Cohen-Boulakia s’étend sur la base de données de l’OMS, qui collecte elle-même partiellement les données des registres. « À cause de l’urgence, on retrouve dans le fichier de l’Organisation mondiale de la santé (OMS) quelques irrégularités qui poussent les médecins à récupérer des informations manquantes dans les registres sources, détaille Sarah Cohen-Boulakia. L’équipe d’Isabelle Boutron a ainsi engagé une véritable armée d’annotateurs pour gérer les problèmes et compléter les informations, ligne par ligne. »
Détecter, trier et comprendre
Mais pour intégrer plus finement les données, en particulier lorsqu’elles sont textuelles, il faut en extraire la substantifique moelle. Pierre Zweigenbaum, directeur de recherche CNRS au Laboratoire d’informatique pour la mécanique et les sciences de l’ingénieur du CNRS, travaille ainsi sur le traitement automatique du langage naturel. Les connaissances médicales ne se trouvent pas que dans des bases de données structurées, mais aussi dans des notes de synthèse et des publications scientifiques. Les bases de données elles-mêmes possèdent des champs en texte libre dont le contenu n’est pas standardisé. Des tâches apparemment simples, comme l’énumération des traitements, posent en fait un certain nombre de difficultés.
Parmi les problèmes de nommage qui peuvent varier entre plusieurs documents, les mots composés sont parfois attachés, d’autres fois séparés, ou encore transformés en acronyme. D’autres embûches compliquent l’automatisation du travail. « En langue naturelle, il existe souvent plusieurs façons différentes d’exprimer la même chose, et inversement, explique Pierre Zweigenbaum. Dans le contexte clinique, l’abréviation “mg” peut aussi bien désigner un milligramme que du magnésium. Lever cette ambiguïté demande de tenir compte du contexte dans lequel le terme est employé. » Avec l’automatisation du traitement du langage naturel, deux types d’erreurs prédominent : détecter trop de termes, ou pas assez. Les chercheurs préfèrent la première option, car il est plus facile de repérer et d’enlever les termes non pertinents que de se rendre compte d’un oubli. Sans cela, des mots inconnus des scientifiques impliqués, comme le mélange de plantes hua-shi bai-du utilisé en médecine traditionnelle chinoise, auraient pu passer à la trappe.
De son côté, l’Institut de recherche en informatique de Toulouse5 a développé des outils d’extraction de notices bibliographiques et un moteur de recherche pour identifier les documents les plus pertinents sur le Covid-19. Ils les collectent dès leur publication en ligne, et indiquent la répartition géographique des recherches menées et les liens de leurs collaborations.
Des outils interactifs pour les chercheurs
Extraites, analysées et agrégées, les informations doivent également être présentées de manière efficace pour faciliter leur interprétation. Romain Vuillemot, maître de conférences au Laboratoire d’informatique en image et systèmes d’information6, travaille ainsi sur la visualisation interactive des données. « Dans le cas du Covid-19, nous avons déployé des programmes de visualisation et de nettoyage des données, afin de déterminer leur qualité et de régler, en collaboration avec les experts du Cress, les anomalies et les erreurs, déclare-t-il. Nous avons identifié 80 % des problèmes lors d’une première phase, puis amélioré nos outils. » Romain Vuillemot a ainsi conçu une carte interactive des études sur le COVID-19, à partir d’une expertise acquise lors de travaux sur les mobilités urbaines.
Des équipes du Laboratoire bordelais de recherche en informatique7 et de l’Institut des systèmes complexes de Paris Île-de-France (CNRS) ont également proposé des solutions afin de regarder les données sous différentes perspectives. Grâce à ça, les médecins explorent plus vite et plus facilement des données complexes réparties sur des tableaux de plusieurs centaines de lignes et d’une centaine de colonnes, et ce sans avoir à être formés à utiliser ces outils. « Les chercheurs du Cress veulent comprendre les liens entre le nombre de cas et les décès du Covid-19, et les traitements employés, souligne Romain Vuillemot. C’est une question très spécifique qui demande de prendre en compte le fait que tous les pays n’ont pas été touchés au même moment. »
Grâce à l’expertise de ces chercheurs, le site fonctionne si bien que l’OMS et plusieurs institutions médicales l’utilisent. « J’ai été impressionnée par le succès de l’appel au CNRS, s’enthousiasme Isabelle Boutron. Tout le monde a mis ses travaux de côté pour nous aider, bénévolement. » ♦
- 1. Unité Inserm/Université de Paris.
- 2. Methods of therapeutic evaluation of chronic diseases, méthodes d’évaluation thérapeutique pour les maladies chroniques.
- 3. Laboratoire de recherche en informatique (LRI, CNRS/Univ. Paris-Saclay), Laboratoire d’informatique pour la mécanique et les sciences de l’ingénieur (Limsi, CNRS), Laboratoire d’informatique en image et systèmes d’information (Liris, CNRS/Univ. Lumière Lyon 2/Centrale Lyon/INSA Lyon/Univ. Claude Bernard Lyon 1), Laboratoire d’informatique, de modélisation et d’optimisation des systèmes (Limos, CNRS/Univ. Clermont-Auvergne/Mines Saint-Étienne), Laboratoire bordelais de recherche en informatique (LaBRI, CNRS/Bordeaux INP/Univ. de Bordeaux), Institut de recherche en informatique de Toulouse (IRIT, CNRS/Toulouse INP/Univ. Toulouse Paul Sabatier), Laboratoire d’informatique de Grenoble (LIG, CNRS/INRIA/Univ. Grenoble-Alpes) et Institut des systèmes complexes de Paris Île-de-France (ISC-PIF, CNRS).
- 4. Unité CNRS/Université Paris-Saclay.
- 5. Unité CNRS/Toulouse INP/Université Toulouse Paul Sabatier.
- 6. Unité CNRS/Université Lyon 2/Centrale Lyon/Insa Lyon/Université Claude Bernard.
- 7. Unité CNRS/Bordeaux INP/Université de Bordeaux.
Voir aussi
Auteur
Diplômé de l’École supérieure de journalisme de Lille, Martin Koppe a notamment travaillé pour les Dossiers d’archéologie, Science et Vie Junior et La Recherche, ainsi que pour le site Maxisciences.com. Il est également diplômé en histoire de l’art, en archéométrie et en épistémologie.