Vous êtes ici
Le vivant a sa matière noire

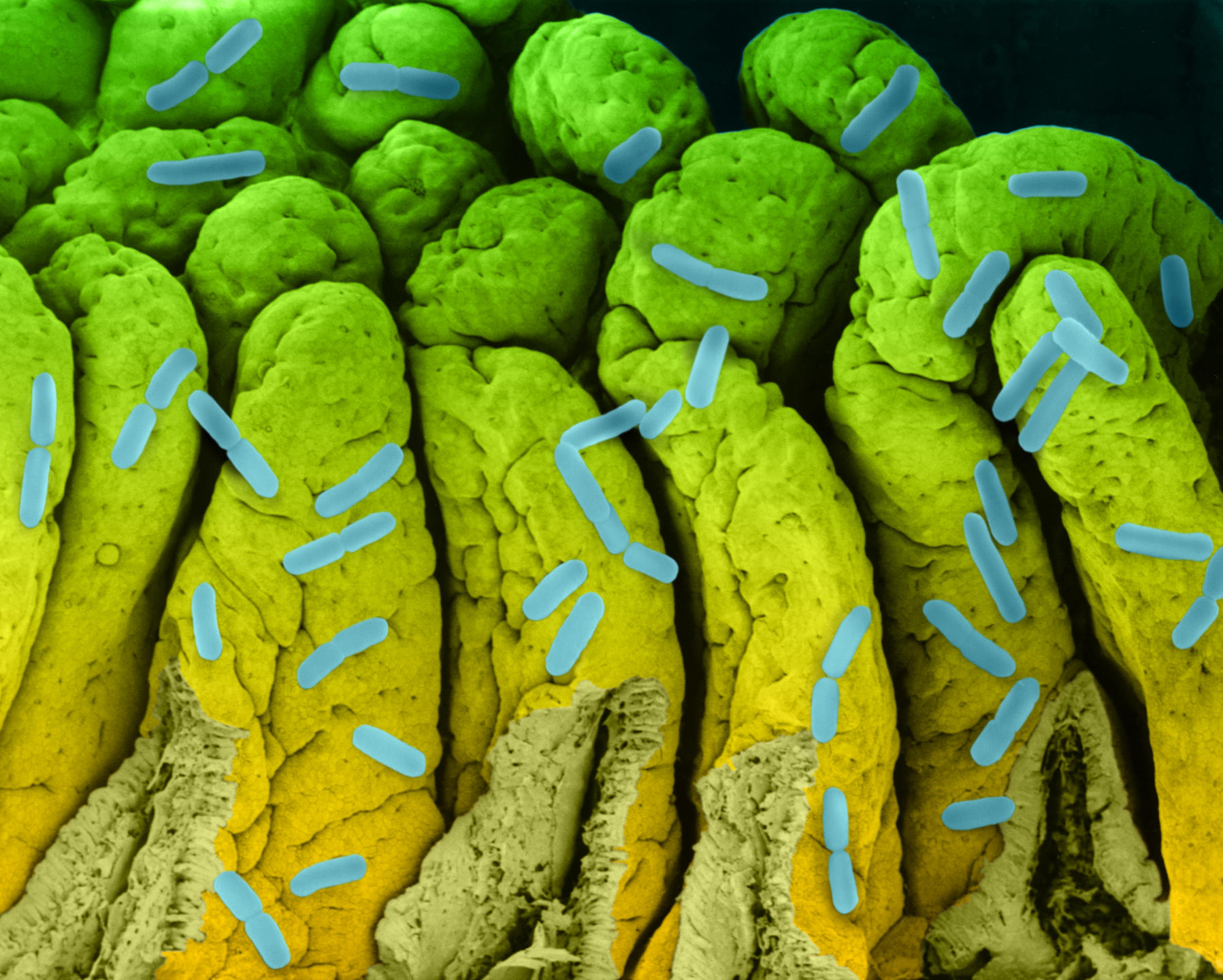
Les biologistes seraient-ils passés à côté de 85 à 99 % des micro-organismes vivant sur Terre ? Les avancées de la métagénomique leur ont en tout cas permis de mettre en évidence l’existence d’une « matière noire » biologique, surnommée ainsi en référence à la mystérieuse matière sombre dont la masse empêche la dislocation des galaxies et des amas galactiques. Un monde biologique qui révèle peu à peu ses secrets et ses surprises.
Trois domaines répertoriés pour le vivant
Les premières classifications du vivant, établies au XVIIIe siècle par Carl von Linné, se basaient principalement sur des critères morphologiques. Ce système, efficient pour de gros organismes tels les mammifères ou les arbres, l’est beaucoup moins dès qu’il s’agit de classer l’ensemble du vivant, micro-organismes compris.
Les chercheurs se sont alors tournés vers des critères moléculaires universels. Ainsi, quelle que soit l’espèce, toutes les cellules biologiques contiennent des ribosomes, des organites qui servent à synthétiser les protéines. Les variations dans les séquences de ces gènes « ribosomaux présents chez tous les organismes vivants » sont utilisées pour évaluer la distance entre espèces et établir les grandes divisions de l’arbre du vivant. C’est notamment en analysant ces gènes chez les procaryotes que, dans les années 1970, Carl Woese a établi la distinction entre le domaine des bactéries et celui des archées.
Les eucaryotes, les bactéries et les archées sont depuis considérés comme les trois domaines du vivant. Les cellules des eucaryotes se caractérisent par la présence d’un noyau et, généralement, de mitochondries. La quasi-totalité des organismes pluricellulaires appartient au domaine eucaryote, qui comprend également des espèces unicellulaires. Bactéries et archées sont dépourvues de noyau, mais ces dernières se distinguent notamment par la composition de leurs membranes.
Un problème de culture
Pour classer la multitude de microbes qu’ils continuent de découvrir, les chercheurs cherchent donc d’abord à séquencer leur génome individuel. Mais cela implique que ces lignées cellulaires puissent être isolées et cultivées, ce qui, finalement, est rarement le cas. Éric Bapteste, chercheur à l’unité Évolution Paris-Seine de l’Institut de biologie Paris-Seine1, rappelle dans sa dernière étude2 qu’au moins 85 % des microbes connus ne sont pas cultivables.
microbes ne vivent
pas seuls rend
la culture pure
difficile.
« Le fait que les microbes ne vivent pas seuls rend la culture pure difficile, explique Éric Bapteste. Ils appartiennent à des collectifs et à diverses formes de symbioses. Parfois, certaines espèces se succèdent dans le temps et il faudrait donc d’abord cultiver leurs précurseurs et reproduire toute la succession. Les chercheurs se trouvent devant un véritable défi biologique, biochimique et temporel. »
Divers outils permettent néanmoins de séquencer une grande partie des gènes présents dans un environnement donné, sans forcément les attribuer à des espèces particulières. On parle de métagénomique. Ces études peuvent aussi bien s’appliquer à notre flore intestinale qu’à des recoins extrêmes de notre planète, comme les geysers.

Les chercheurs inventorient les séquences génétiques obtenues dans ces analyses métagénomiques, puis en retranchent celles qui correspondent aux organismes connus et cultivés. Pour explorer la diversité des séquences génétiques, l’équipe d’Éric Bapteste et de son collègue Philippe Lopez a employé une méthode en deux temps. Ils ont cherché dans l’environnement des séquences ressemblant à celles déjà connues, puis ont ensuite effectué une seconde analyse pour établir des liens supplémentaires.
« Si l’on prend l’exemple d’une recherche de parenté à partir de photos de famille, précise Éric Bapteste, la ressemblance entre un individu et son grand-père ne sera pas forcément évidente. Des liens peuvent cependant être trouvés si l’on rapproche l’enfant de son père, puis son paternel de son grand-père. Le lien indirect entre les individus de ces trois générations apparaît alors. »
A-t-on découvert un quatrième domaine ?
Une partie des très nombreuses séquences qui restent non attribuées pourraient provenir d’espèces nouvelles incultivables. Certaines séquences sont particulièrement intrigantes. Bien qu’elles se rattachent à des familles de gènes connus, elles en divergent tellement qu’on a pu se demander si elles ne provenaient pas d’organismes eux-mêmes très divergents, appartenant à un quatrième domaine inconnu du vivant.
« Il revient aux systématiciens de dire à quel moment une forme de vie doit rentrer dans une nouvelle case, modère toutefois Éric Bapteste. Les chercheurs se basent sur des critères métaboliques, structuraux ou génétiques, mais il n’existe pas de recette fixe pour affirmer l’existence d’un nouveau domaine. Le processus n’a rien d’automatique, il réclame des arguments et un consensus parmi les chercheurs. À ce stade, nous n’avons aucune raison de dire qu’un nouveau domaine a été découvert. »
peuvent évoluer
très vite. Ils se
retrouvent alors à
une place étrange
dans un arbre
phylogénétique.
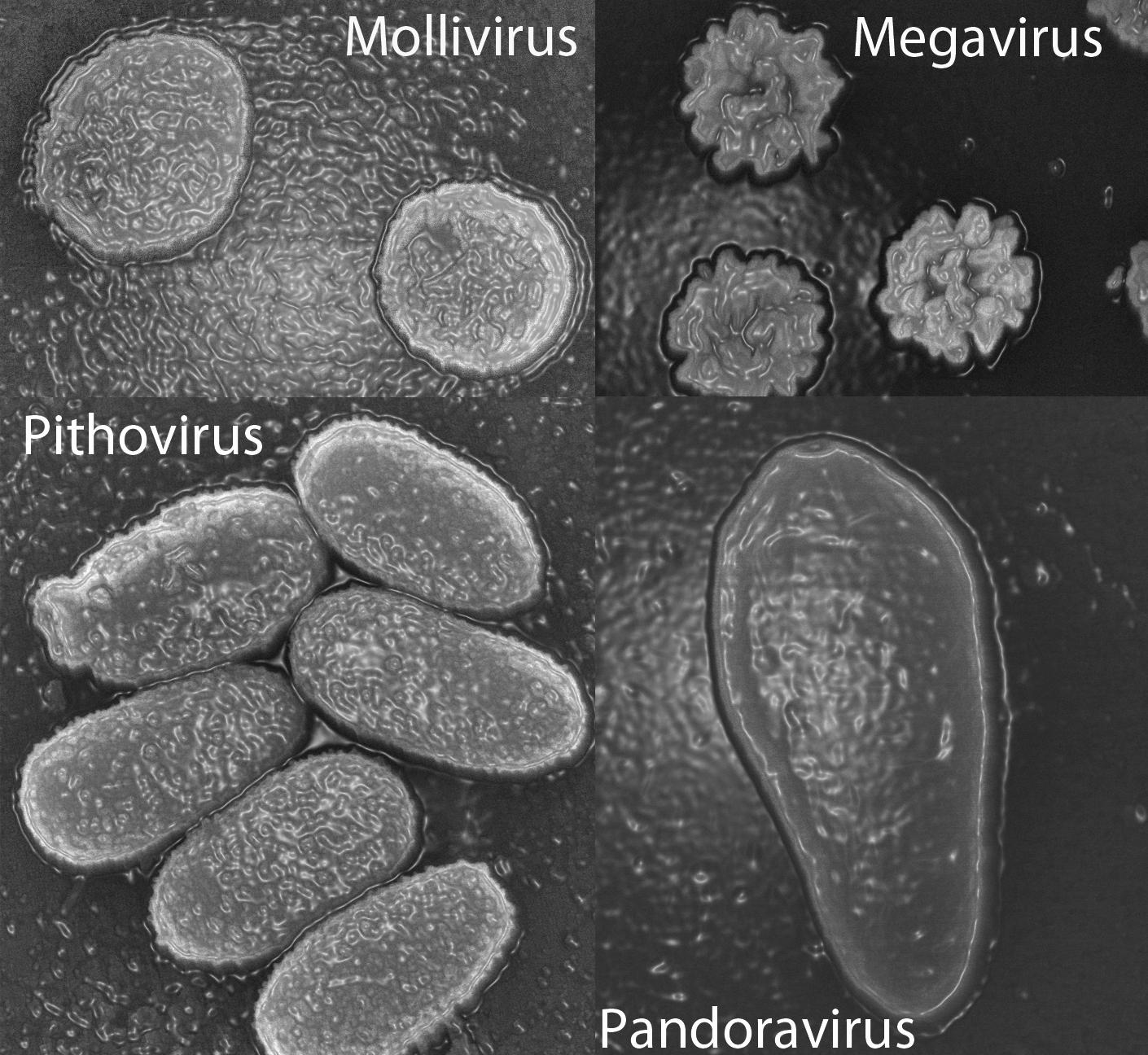
Le même type d’interrogation avait suivi la découverte en 2013 de deux virus géants, Pandoravirus salinus et Pandoravirus dulcis. En effet, seuls 7 % des gènes de ces virus correspondaient à des séquences connues, et ces derniers étaient dépourvus de capside, la structure qui entoure normalement le matériel génétique des virus.
Purificación Lopez-Garcia, chercheuse au laboratoire Écologie, systématique et évolution3, estime que les analyses phylogénétiques n’ont pas permis d’étayer l’appartenance des virus géants à un quatrième domaine du vivant.
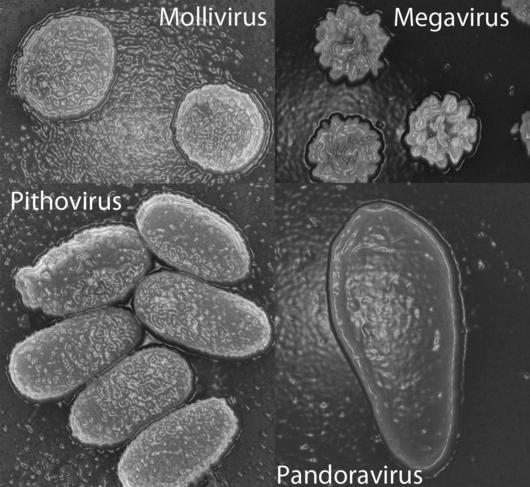
« Il peut être tentant d’affirmer que certains gènes forment un quatrième domaine, prévient Purificación Lopez-Garcia, mais certains peuvent évoluer très vite, par exemple à cause des duplications géniques, à la suite de transferts horizontaux ou de leur capture par un génome viral. Ils se retrouvent alors à une place étrange dans un arbre phylogénétique. Cela n’implique pas que l’organisme auquel ils appartiennent constitue pour autant une espèce complètement à part. On ne peut rien dire tant que les génomes n’ont pas été reliés à des organismes précis ; or, pour l’instant, tout ce qui a été lié à un microbe cultivé ou à un organisme reconstruit à partir de métagénomes rentre bien dans les trois domaines du vivant. »
Une matière noire aux effets visibles
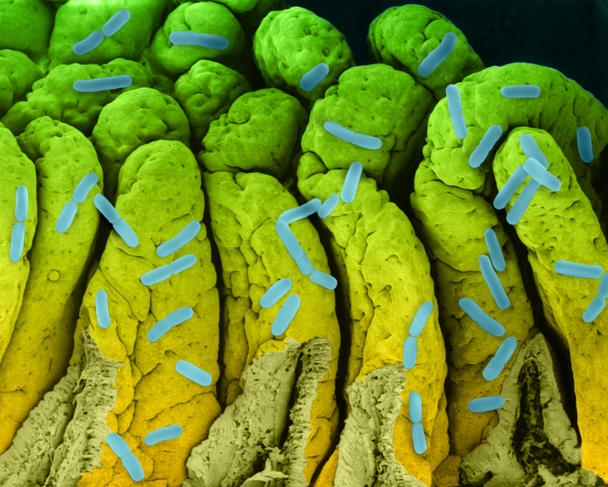
Si elle n’a pas débouché sur l’inauguration d’un quatrième domaine de la vie, l’étude de la matière noire biologique a en tout cas produit de très beaux fruits. Dusko Ehrlich, directeur de recherche émérite au projet MetaGenoPolis de l’Institut national de la recherche agronomique et directeur du Centre d’interaction entre hôte et microbiome du King’s College de Londres, a ainsi travaillé sur la diversité de notre flore intestinale. Il souligne les énormes progrès réalisés depuis 2008 au sein du projet européen MetaHIT, consacré à la métagénomique.
Une première étude publiée en 20104, portant sur 124 individus européens, a ainsi inventorié 3,3 millions de gènes microbiens intestinaux, soit 150 fois plus que les 23 000 gènes de notre génome. Lorsque l’équipe a augmenté sa cohorte à 1 200 personnes, c’est 9,9 millions de gènes qui ont été identifiés. De cette diversité génétique, 750 groupes de plus 700 gènes bactériens ont été identifiés, et 85 % des espèces de bactéries se sont révélées nouvelles.
Ces travaux ont permis de caractériser le microbiome intestinal de l’homme. On a pu ensuite comparer les microbiomes de personnes malades et saines. Un lien a ainsi été découvert entre la composition de certains microbiomes et le risque de cirrhose5. Cela a débouché sur une méthode de diagnostic fiable à 95 % basée sur le séquençage des selles. Une avancée qui pourrait remplacer la très invasive biopsie hépatique.
représentent
en chacun de nous
une masse qui
peut-être plus
importante
que le cerveau.
Une autre étude6 a montré que la perte de diversité du microbiome intestinal est associée à une prédisposition au diabète, aux maladies hépatiques, à certains cancers et aux maladies cardiovasculaires. Or sept de ces maladies entrent dans les dix causes les plus fréquentes de mort dans le monde.
« Le microbiome intestinal fonctionne comme un organe négligé, déplore Dusko Ehrlich. Ces bactéries représentent en chacun de nous une masse qui peut-être plus importante que le cerveau. Pourtant, le grand public ne se rend pas compte de sa vulnérabilité. Il jugerait pourtant absolument catastrophiques de telles atteintes à ses autres organes. »
Qu’elle se cache dans le sol, dans les océans ou à l’intérieur de nos intestins, le rôle biologique joué par la matière noire se révèle donc capital tant du point de vue quantitatif – sa masse totale semble colossale – que qualitatif – de par sa contribution à l’équilibre des écosystèmes, quelle que soit l’échelle considérée. L’éclairage nouveau permis par la métagénomique promet en tout cas de dévoiler une diversité biologique aux ramifications jusqu’ici insoupçonnées.
Lire aussi: Les microbes, pilotes de nos conflits intérieurs?
- 1. Unité CNRS/UPMC/Inserm.
- 2. « Highly divergent ancient gene families in metagenomic samples are compatible with additional divisions of life », Lopez et al., Biology Direct, 26 octobre 2015, vol. 10 : 64.
- 3. Unité CNRS/Univ. Paris Sud/AgroParisTech.
- 4. « A human gut microbial gene catalogue established by metagenomic sequencing », Qin et al., Nature, 4 mars 2010, vol. 464 : 59-65.
- 5. « Alterations of the human gut microbiome in liver cirrhosis », Qin et al., Nature, 4 septembre 2014, vol. 513 : 59-64.
- 6. « Richness of human gut microbiome correlates with metabolic markers », Le Chatelier et al., Nature, 29 août 2013, vol. 500 : 541–546.
Voir aussi
Auteur
Diplômé de l’École supérieure de journalisme de Lille, Martin Koppe a notamment travaillé pour les Dossiers d’archéologie, Science et Vie Junior et La Recherche, ainsi que pour le site Maxisciences.com. Il est également diplômé en histoire de l’art, en archéométrie et en épistémologie.