Vous êtes ici
Les nouvelles frontières du vivant
Temps de lecture : 14 minutes
Depuis quand la biologie « moderne » existe-t-elle ?
Catherine Jessus1. La biologie telle qu’on la pratique aujourd’hui est relativement récente. Elle résulte de la fusion, à la fin du XIXe siècle, de deux grands courants nés dans l’Antiquité : les naturalistes et les médecins physiologistes. Le courant des naturalistes, dont Aristote est l’un des premiers représentants, avec plus près de nous des savants comme Linné, Lamarck ou même Darwin, n’a eu de cesse d’observer, de décrire et d’inventorier méthodiquement le vivant. On a l’habitude de situer les débuts du naturalisme dans l’Antiquité, mais si l’on regarde les grottes de Lascaux et de Chauvet, on voit dès la préhistoire des représentations d’animaux d’une précision et d’une justesse qui font de nos lointains ancêtres des observateurs aiguisés de la nature qui les entoure.
Le deuxième courant, celui des physiologistes liés à la médecine, né avec Hippocrate et Galien et incarné au XIXe siècle par un scientifique comme Claude Bernard, s’attache lui à comprendre le fonctionnement du corps humain : à quoi sert le sang, comment le cœur marche, etc. Ces deux courants se rejoignent à l’orée du XXe siècle lorsque les naturalistes s’emparent des méthodes expérimentales des physiologistes pour les appliquer à la zoologie et à la botanique. Cela marque l’essor de la science biologique à proprement parler, qui n’est plus seulement dans l’observation mais place l’expérimentation au cœur de sa démarche intellectuelle : on pose une hypothèse et on développe un protocole expérimental pour répondre à la question.
Qu’est-ce qui a permis les grandes avancées de cette toute nouvelle science biologique au XXe siècle ?
C. J. Le moteur essentiel repose sur de formidables progrès en termes d’outils. Les pas de géant faits par l’imagerie et la microscopie durant tout le XXe siècle ont permis de voir jusqu’à l’intérieur de la cellule elle-même. Il fallait néanmoins découper le matériel biologique en « tranches » pour pouvoir le mettre sous le microscope, c’est-à-dire commencer par le tuer avant de l’observer. Aujourd’hui, avec l’usage des marqueurs fluorescents, les biologistes ont la possibilité d’étiqueter une molécule et de la suivre à l’intérieur d’un organisme vivant. L'’avènement de la biologie moléculaire dans les années 1950 a livré des connaissances fondamentales sur le génome, sa structure, son fonctionnement, et ouvert la voie à sa manipulation.
Avec l'arrivée ces vingt dernières années de la méta-génomique, nous avons désormais la vision de l'ensemble des génomes contenus dans un échantillon prélevé dans l'environnement – que ce soit un échantillon d'eau de mer, de rivière, de sol, voire dans le contenu intestinal d'un être vivant – une petite révolution permise par l'avènement du big data (le traitement des données de masse) qui lève le voile sur l’infinie diversité du monde microbien dont nous connaissons si peu de choses.
J’ajouterai à ces avancées les progrès très récents faits en ingénierie du génome grâce à des outils comme les ciseaux génétiques Crispr-Cas9, notamment, mais aussi en ingénierie tissulaire et cellulaire qui permet d’étudier le fonctionnement d’une cellule unique. La liste des avancées récentes est impressionnante, et pourtant : le champ de ce qu’il nous reste à découvrir ne cesse de s’agrandir...
Est-ce à dire que, plus on en sait sur le vivant, plus l’on se rend compte que l’on connaît peu de lui ?
C. J. C’est à peu près ça, oui. Prenez l’exemple de la molécule d’ADN découverte dans les années 1950. Chez l’homme, notre ADN fait 2 mètres de long – 2 mètres qui se logent dans un noyau cellulaire de 5 microns. Outre le défi de faire rentrer un tel contenu dans un si petit contenant, il faut réaliser que sur ces 2 mètres, seuls 6 centimètres sont dits codants, c’est-à-dire portent les gènes qui codent pour la fabrication des protéines. Tout le reste est composé d’ADN non codant – qu’on a appelé « ADN poubelle » jusqu’à ce que l’on se rende compte, il y a quelques années, qu’il est transcrit en ARN non messagers dont on commence tout juste à entrevoir l’importance... Ces ARN aident par exemple les plantes à lutter contre les infections virales ou bactériennes, et c’est un grand ARN non codant qui enveloppe l’un des deux chromosomes X de la femme afin de bloquer son expression.
Quels sont, alors, les grands enjeux pour les biologistes du XXIe siècle ?
C. J. Je citerais l’exploration du monde microbien, qui inclut des organismes vivant dans des milieux extrêmes et de nouvelles formes de vie inattendues ; l’étude des moteurs de l’évolution, au-delà des seules mutations génétiques ponctuelles ; le fonctionnement des génomes, dont nous venons juste de parler ; l’étude des interactions entre êtres vivants, et entre êtres vivants et environnement - étudier des organismes isolément perd de son sens quand on sait que de la bactérie à l’humain en passant par les plantes, les êtres vivants sont tous intégrés dans des systèmes complexes à l’équilibre fragile ; enfin, les fonctions cognitives et le cerveau, mais aussi le système immunitaire, un trop vaste sujet que nous ne pouvons développer dans le cadre de notre entretien. Et encore, cette liste est loin d’être exhaustive !
Commençons par la définition de ce qui fait la vie même… Vous nous dites qu’elle a changé ces dernières années. Qu’est-ce qui a conduit à cette remise en perspective ?
C. J. Jusqu’au début du XXIe siècle, on considérait que le vivant ne pouvait se développer dans des milieux de vie qualifiés d’extrêmes : au-delà de certains seuils de température, de pression, de salinité..., en l’absence de lumière ou d’oxygène, la vie était jugée impossible. La découverte d’organismes vivants dans des environnements extrêmes – dans des lacs ou des geysers bouillants, dans l’obscurité des abysses ou dans des milieux acides comme le site volcanique de Dallol en Éthiopie – oblige à reposer la question « qu’est-ce que le vivant ? ».
Ces organismes extrémophiles, des micro-organismes pour l’essentiel, ont des métabolismes, des membranes cellulaires et d’autres caractéristiques que l’on ne pouvait pas imaginer. Ils devraient également pouvoir nous éclairer sur les origines de la vie sur terre, il y a 3,8 milliards d’années, et sur ce que nous avons à chercher sur d’autres planètes... En attendant, étudier les êtres vivants de ces milieux extrêmes pose de vrais défis techniques aux chercheurs : pour l’instant, on est en effet incapable de cultiver la plupart de ces formes de vie en laboratoire.
Venons-en au monde microbien, justement, dont vous nous dites qu’il est une véritable terra incognita...
C. J. Le monde microbien continue de nous échapper, même dans des environnements plus proches et familiers que les milieux extrêmes que nous venons d’évoquer. Grâce à l’expédition Tara océans conçue par le biologiste Eric Karsenti, on a notamment découvert qu’une myriade de micro-organismes peuplent les océans : ainsi, on estime que 60 % des bactéries présentes sur notre planète sont dans les mers, or on en connaît à peine 5 % ! Il reste un énorme travail de description et d’inventaire à mener, proche de ce qu’ont pu réaliser les naturalistes du passé. Il faut aussi changer de regard sur ces micro-organismes : on a tendance à les voir uniquement comme des pathogènes – pourtant, seule une infime minorité de bactéries et de virus est nocive pour l’homme -, alors qu’ils jouent bien au contraire un rôle majeur dans l’équilibre de la planète, qu’on commence seulement à entrevoir.
Imaginez que cela fait quelques dizaines d’années à peine qu’on a découvert le règne des archées, la 3e branche du vivant à côté des bactéries et des eucaryotesFermerLes eucaryotes sont les organismes unicellulaires ou multicellulaires dont les cellules contiennent un noyau. Très présentes dans les environnements extrêmes, les archées se retrouvent en réalité dans tous les écosystèmes. Or nous les connaissons très peu et commençons juste l’exploration de leur génome, très complexe et doté de nombreux gènes aux fonctions inconnues… Et que dire des virus géants découverts au tournant du XXIe siècle !
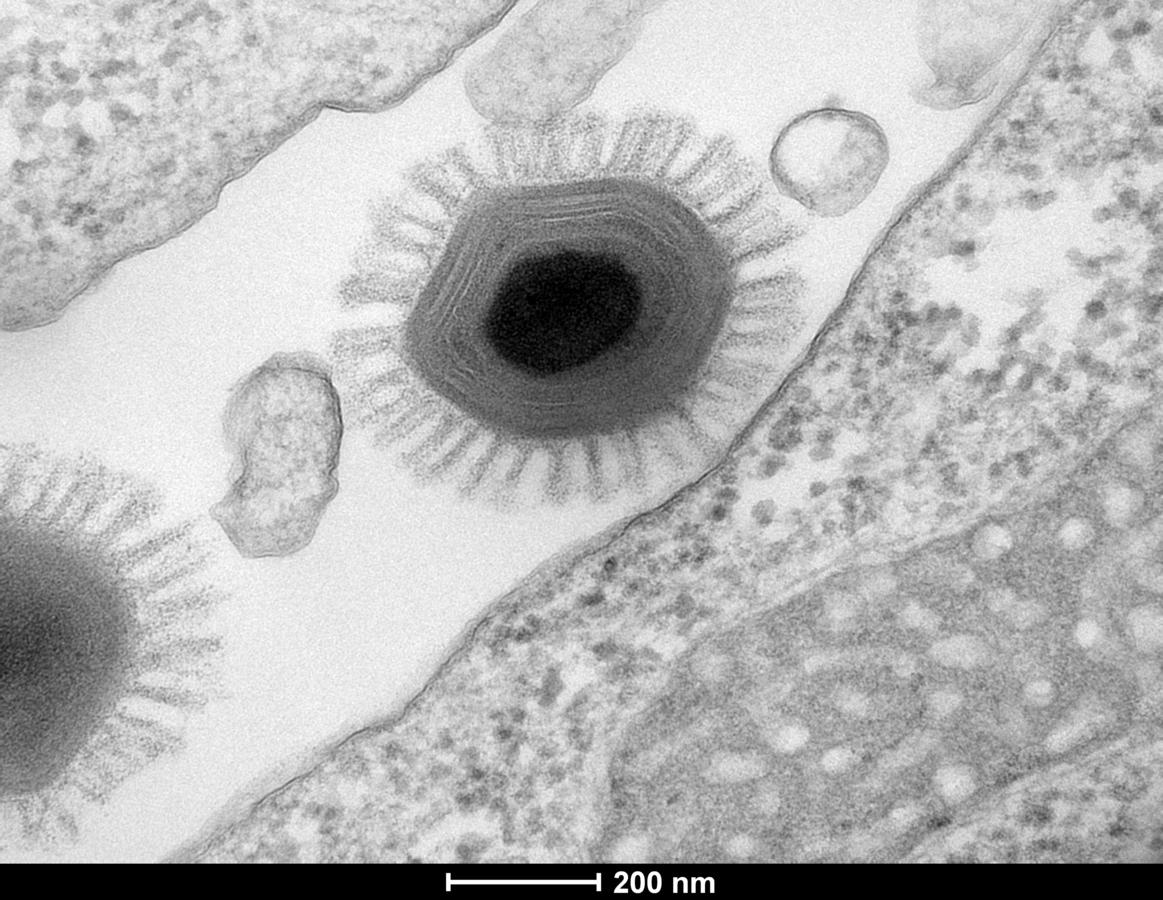
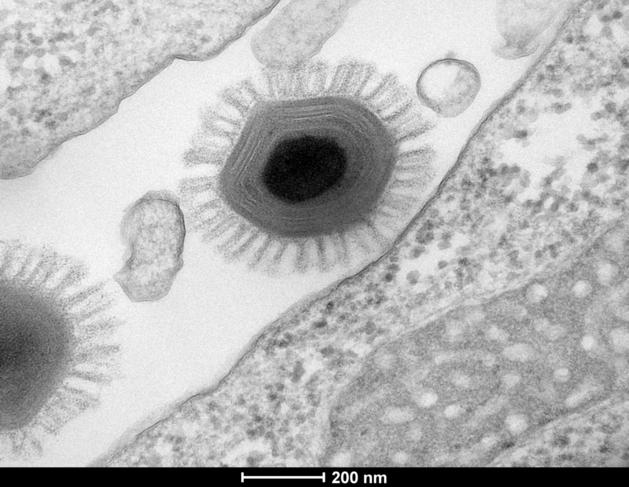
Que sait-on de ces virus géants ?
C. J. Leur appellation de « virus » est en réalité abusive. Comme les virus « classiques », ce sont des parasites qui ne peuvent se multiplier hors d’une cellule-hôte. Mais contrairement aux virus habituels, ils ont des génomes énormes et complexes, composés de milliers de gènes, une taille énorme, de celle d’une bactérie, et une enveloppe très particulière. D’où viennent-ils ? Comment les qualifier, eux qui ne sont ni des virus ni des cellules ? Les discussions sur le sujet sont nourries. Toujours est-il qu’on en retrouve partout sur la planète, logés dans les amibes qui peuplent toutes les eaux, comme celle du robinet. A priori, ils ne sont pas pathogènes, mais nous sommes très loin de les connaître tous ! Et qu’en est-il des mégavirus emprisonnés dans le pergélisol depuis près de 30 000 ans, que le réchauffement climatique est en train de libérer progressivement du fait du dégel de ces sols gelés, et avec lesquels l’homme actuel n’a pas coévolué ?
Qu’en est-il des moteurs de l’évolution ? Pourquoi est-il important de s’y pencher dans les décennies à venir ?
C. J. On a longtemps pensé que c’étaient les mutations ponctuelles de gènes qui permettaient de créer de nouvelles espèces. Mais ce n’est en réalité qu’un outil parmi d’autres pour créer du nouveau. La principale source d’innovation vient de la modification de vastes pans de génomes, permis par les échanges ou les fusions de génomes entre espèces, par exemple.
On sait ainsi que c’est l’association d’une archée et d’une bactérie qui a permis de « fabriquer » la première cellule eucaryote. La bactérie qui s’était introduite dans l’archée a progressivement perdu son autonomie et a permis la genèse des compartiments membranaires, en particulier la mitochondrie, qui caractérisent les eucaryotes. Même chose pour l’apparition des plantes : c’est un eucaryote entré en symbiose avec une bactérie capable d’utiliser la lumière pour fabriquer de l’énergie, qui a donné naissance à la première micro-algue. C’est encore ce même mécanisme qui explique pourquoi les mammifères ont un placenta qui permet à l’embryon de se développer à l’intérieur même de l’individu maternel.
Les mammifères sont eux aussi le fruit d’un échange de gènes ?
C.J. Exactement. Le placenta a pu se développer grâce à l’apport de gènes d’un rétrovirus à un animal qui pondait des œufs. Ce rétrovirus particulier était capable de produire des protéines occasionnant des fusions cellulaires – lesquelles protéines sont à l’origine du placenta. Sans ce petit rétrovirus, nous pondrions encore des œufs ! Ce type d’exemples nous montrent combien les échanges de matériel génétique entre êtres vivants – longtemps négligés - sont importants.
Les interactions : le dernier grand défi de la biologie des prochaines années…
C. J. Les associations sont nécessaires à la vie. Tout être vivant est soit contenu dans un être plus grand, soit contient des êtres plus petits – aucun n'existe de manière isolée. Le microbiote intestinal en est une bonne illustration : composé d’archées, de bactéries, de virus et de levures, il peut peser jusqu'à 2 kg et fonctionne comme l’un de nos organes - ce qui pose au passage de vraies questions sur ce qui est le « soi » et le « non soi ». Ce microbiote est indispensable au fonctionnement de notre système digestif - sans lui, on ne pourrait dégrader tous les aliments qu’on ingère -, mais il permet aussi la maturation de notre système immunitaire et sécrète des molécules cruciales qui agissent sur le fonctionnement de nos organes, dont notre cerveau !
Tout déséquilibre dans ce système est source de perturbations : ainsi une bactérie du microbiote qui se met à proliférer, profitant de l’affaiblissement d’autres espèces microbiennes, peut très vite devenir maligne. En réalité, nous sommes en train de découvrir que le vivant est un monde complexe où tout est question de dose et d’équilibre, que ce soit à l’échelle d’un individu, d’une population ou de tout un écosystème. C’est cette complexité que nous devons aujourd’hui aborder. Le temps où l’on étudiait de façon isolée telle cellule, tel tissu, tel organe, tel individu..., est révolu : les biologistes doivent étudier aujourd’hui des réseaux constitués de myriades d’acteurs.
Le dernier exemple que je donnerai, et sans doute le plus spectaculaire, est celui du cerveau, un organe d’une complexité folle par le nombre des cellules impliquées. Là, tout n’est que réseaux et interactions. Grâce aux données de l’imagerie, on commence à en avoir une bonne cartographie et à comprendre quelle zone interagit avec quelle autre, à comprendre comment circule l’information électrique au niveau nanométrique de la synapse. Mais le vrai chalenge pour les biologistes reste le cerveau cognitif : la mémoire, le souvenir, les émotions, la prise de décision, et toute la chimie qui pilote ces fonctions. Ce qu’il nous reste à découvrir, là encore, est vertigineux. ♦
Événement
Lancement de l'Année de la biologie, le 19 octobre. Toutes les informations sur le site dédié à cette année.
À lire sur notre site
De l'extraordinaire diversité de la vie
- 1. Directrice de recherche CNRS au Laboratoire de biologie du développement (CNRS-Sorbonne Université), où elle dirige l’équipe Biologie de l’ovocyte. Elle a dirigé l’Institut des sciences biologiques du CNRS de 2013 à 2019.
Voir aussi
Auteur
Journaliste scientifique, Laure Cailloce est rédactrice en chef adjointe de CNRS Le Journal et de la revue Carnets de science.